地理空間情報を扱う上で知っておきたいPythonライブラリ、GeoPandas入門~応用編・衛星データと組み合わせ~
地理空間情報を解析する上で有用なPythonライブラリGeoPandas。後編では、衛星データとシェープファイルを組み合わせて解析することで、テーブルデータを作成することにチャレンジします。筆ポリゴンを任意の大きさのベクターデータで切り取り、切り取ったデータを用いて、衛星データをその範囲で取得し、NDVIの推移を求めます。
前編ではシェープファイルのような地図に重ねることのできる幾何的な情報を含んだデータと、単なるテーブルデータ(ただし、そのシェープファイルと同様のデータを含む場合。例えば都道府県の名称)を重ねて、作成したデータを描画するということを学びました。
後編となる本記事では、シェープファイルと同様に幾何的なデータを含みつつ、ラスターデータとして扱われているもの(代表的なものだとGeoTIFF)を、シェープファイルを始めとしたベクターデータで切り取りテーブルデータにしてみましょう。
上の作業は衛星データ解析にはお馴染みですが、今回はより農業寄りの話となっています。というのも、日本には筆ポリゴンという耕作地の区画を示したシェープファイルがあります。こちらは200メートル四方の区画で全国の土地を区切ったもので、市町村レベルであったとしても対象となる地物の数は膨大なものになります。そのため、そのまま利用することは難しいです。このような場合にはベクターを切り抜きを行うことで対処をします。
今回は、ある自治体の筆ポリゴンを任意の大きさのベクターデータで切り取り、切り取ったデータを用いてさらに衛星データをその範囲で取得することにします。
本記事もGoogle Colaboratoryで動作しますので、プログラムを実行したい方はこちらも合わせてご覧ください。
この章で利用するデータは以下の通りです:
前編と同様にライブラリをインポートし、解析の準備を進めます。
# Important library for many geopython libraries
!apt install gdal-bin python-gdal python3-gdal
# Install rtree - Geopandas requirment
!apt install python3-rtree
# Install Geopandas
!pip install git+git://github.com/geopandas/geopandas.git
# Install Folium for Geographic data visualization
# !pip install folium
!pip install plotly-express
!pip install --upgrade plotly
!pip install matplotlib-scalebar
# Use EE in Python
!pip install geemap
!pip install ipygee# Colab使用時
import os
os.kill(os.getpid(), 9)# Driveのマウント
# Filesからもワンクリックでマウント可能です
from google.colab import drive
drive.mount('/content/drive')ライブラリのインポートを行います。全てがインストールされていれば、エラーなく実行できます。
import pandas as pd
import numpy as np
import os
import geopandas as gpd
from shapely.geometry import Point
import matplotlib
import matplotlib.pyplot as plt
import folium
import plotly_express as px
from datetime import datetime
import geemap
from ipygee import*衛星データ(ラスターデータ)との組み合わせ
ここからは、衛星データを組み合わせた解析例を見てみましょう。
- ・eeを用いた衛星データ(MOD13Q1)の読み込み
- ・ベクターデータを用いた画像の切り取り
- ・可視化
を行います。
今回は自作のベクターデータで行っていますが、これを筆ポリゴンのような耕作地を表現するベクターデータと組み合わせることによって、収穫量の予測を行うことが可能になります。
座標系の変換
座標系といえば、誰もが知るxy座標です。こちらは直行座標と呼ばれるもので互いに直行した座標により点の位置が定まるものです。地理空間情報では、三次元の座標系を基本とし、それを平面に投影するということを行っています(下図)。
座標参照系については、様々なところで解説を見ることができます。ここでは簡単に、座標参照系とは地理座標系(Geographic Coordinate System)と投影座標系(Projected Coordinate System)からなるものだと覚えておきましょう。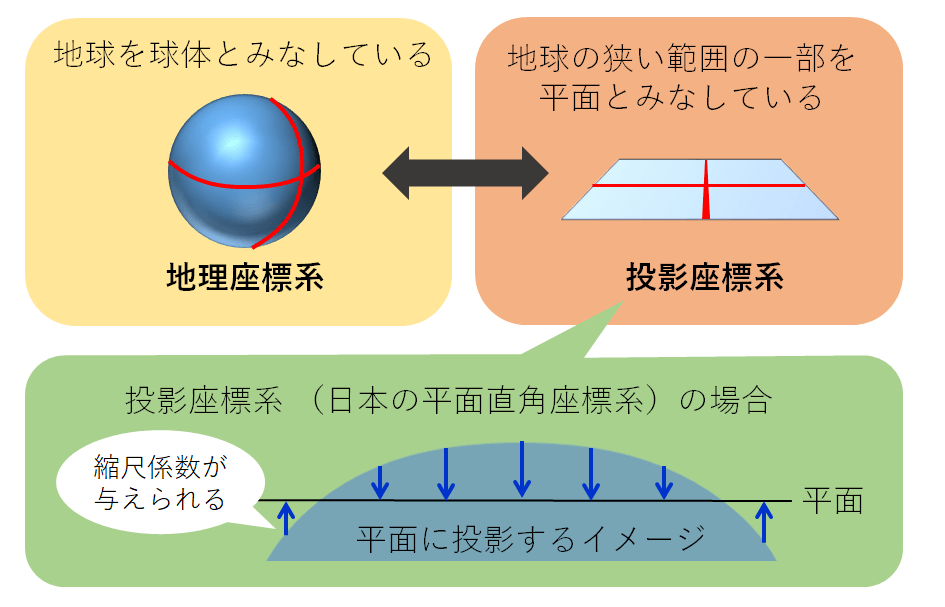
簡単な理解としては、地理座標系とは緯度や経度のようなもので、対象の位置を示したものです。一方で投影座標系では球体を平面に置き換え、距離で対象の位置を示したものです。そのため、ある地物(ベクターデータで表現される一つ一つのオブジェクト)の面積を計算したい場合には投影座標系を利用するのが便利になります。
# read GeoJSON
# ファイルを保存する階層に合わせて適宜パスは変更してください
shounai = gpd.read_file('/content/drive/MyDrive/Sorabatake/shounai.geojson')
shounai.head()shounaiの空間参照系を確認しましょう。
shounai.crsName: WGS 84
Axis Info [ellipsoidal]:
– Lat[north]: Geodetic latitude (degree)
– Lon[east]: Geodetic longitude (degree)
Area of Use:
– name: World.
– bounds: (-180.0, -90.0, 180.0, 90.0)
Datum: World Geodetic System 1984 ensemble
– Ellipsoid: WGS 84
– Prime Meridian: Greenwich
WGS84とはWorld Geodetic System(世界測地系)1984の略です。WGS84は、米国が構築・維持している世界測地系となります(下図)。
測地系は座標系のベースとなるもので、これが決まらなければ座標を定めることはできません。測地系にも様々な種類があり、世界全体の位置関係を示すものや、局地的に特化した位置関係を捉える測地系があります(特化しているというのは必ずしも精確という意味ではないです)。
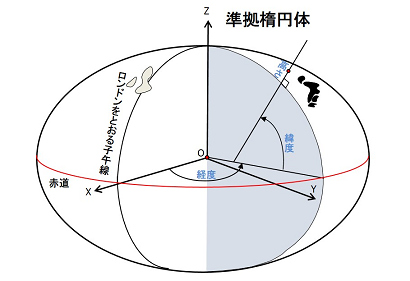
地球は凹凸のある楕円体ですが、測地系では、その凸凹を理想的な楕円体に当てはめて滑らかにし、これにより対象の位置関係を定義しています。詳しくはこちらの記事をご参照ください。
foliumで簡単に場所を示してみます。
下のセルでは、geometryというメソッドを利用しています。GeoSeriesの属性はメソッドはほとんどがshapely由来のものです。
latlon = [shounai.geometry.centroid.y[0],shounai.geometry.centroid.x[0]]
m = folium.Map(latlon, zoom_start=12, control_scale=True)
folium.GeoJson(shounai).add_to(m)
m
GeoSeriesの属性(Attributes)として以下があります:
- – area
- – bounds
- – total_bounds
- – geom_type
- – is_valid
一つずつ見てみましょう。
# areaでは地物の面積を出すます。つまり一つ一つのgeometryが持っている面積になります
# 指定されたCRSによっては正しく面積を出すことができません。
# 正しい投影座標を選ぶことが大切です(後述します)
shounai.area# boundsではx方向、y方向における最大値と最小値を出します
# これらの点を使えば、対象となるgeometryを包み込む四角形が描けます。衛星画像をダウンロードする際に指定する際に使ったりする点です。
# stackoverflowの例で見ると分かりやすいです。
# https://gis.stackexchange.com/questions/254815/simple-way-to-collect-the-sentinel-data-of-a-given-roi
shounai.bounds補足ですが、envelopeを利用しても同じことが可能です。envelopeでは、geometryを包む四角形を作りだすことができます。
shounai.geometry.values[0]
shounai.geometry.values[0].envelope
# こちらは全てのgeometryから見た最小値と最大値になります。
# envelopeの点はこちらを参照しています。
shounai.total_boundsarray([139.78055477, 38.7860372 , 139.80209827, 38.81212507])
# geometryのタイプを見ます
# 上で示したように、ポリゴンやポイントなどがこれにあたります。
shounai.geometry.geom_type0 Polygon
dtype: object
# geometryの形状が正しいものかの確認が行えます
# 自分で作成した、また信頼できる期間が作成したベクターファイルであれば、ここが不正なデータになることは少ないと思います
# 仮に不正なデータがあったとすれば、QGISでその対象の地物を調べてみるのが良いと思います。GeoPandasではこの不正な地物を修正することは難しいです
shounai.is_valid0 True
dtype: bool
それでは取得したデータの空間参照系を確認してみましょう。
shounai.crsName: WGS 84
Axis Info [ellipsoidal]:
– Lat[north]: Geodetic latitude (degree)
– Lon[east]: Geodetic longitude (degree)
Area of Use:
– name: World.
– bounds: (-180.0, -90.0, 180.0, 90.0)
Datum: World Geodetic System 1984 ensemble
– Ellipsoid: WGS 84
– Prime Meridian: Greenwich
EPSG4326となっています。
EPSG(European Petroleum Survey Group)は各国の様々な測地系や投影法に対して、固有のID番号が与えられたものです。EPSG4236はWGS84に該当します。
該当のデータがWGS84の参照系を利用していることがわかります。今回は、作成したベクターデータの面積とそこに含まれる衛星画像の値を抽出したいので、この地理座標系を投影座標系に置き換える必要があります。
この場合、UTM(Universal Transverse of Mercator)を利用するのが便利です。GeoPandasの基本的なメソッドであるto_crsを利用して参照系の変換をしましょう。
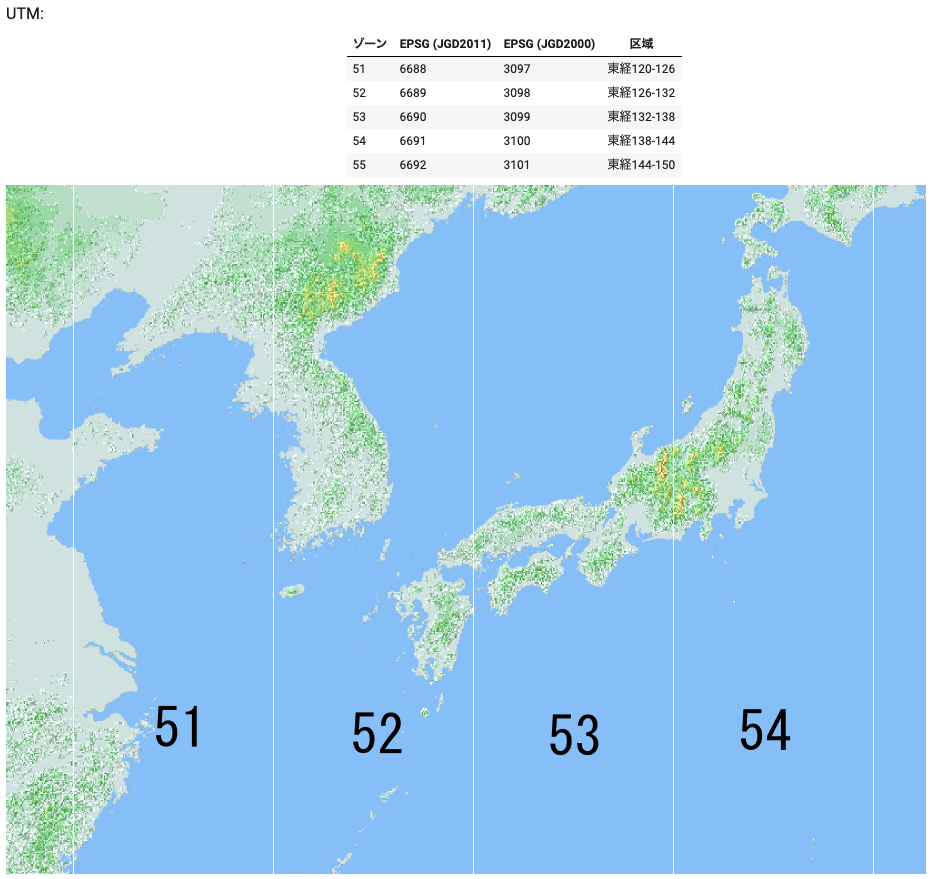
山形はUTM54に当たるため、EPSG 6691を指定すれば良いことがわかります。
# Convert 4326 to 6691
shounai.to_crs('epsg:6691',inplace=True)# Check its crs
shounai.crsName: JGD2011 / UTM zone 54N
Axis Info [cartesian]:
– E[east]: Easting (metre)
– N[north]: Northing (metre)
Area of Use:
– name: Japan – between 138°E and 144°E, onshore and offshore.
– bounds: (138.0, 17.63, 144.0, 46.05)
Coordinate Operation:
– name: UTM zone 54N
– method: Transverse Mercator
Datum: Japanese Geodetic Datum 2011
– Ellipsoid: GRS 1980
– Prime Meridian: Greenwich
WGS84からJGD2011測地系に置き換わっているのが分かります。投影座標系はUTM 54になっています。(Datumというのは測地系)。
それでは、面積(ヘクタール)を計算してみましょう。
shounai['area_ha'] = shounai.geometry.area/10**4
shounai.head()約172ヘクタールであることがわかりました。
二つのベクターデータの結合
衛星画像データのようなラスターデータはそのまま使うにはあまりに範囲が広すぎることがよくあります。そのため基本的に関心領域と呼ばれるベクターデータで範囲を制限します。しかしベクターデータがそもそも扱い難い場合にどうでしょうか?
例えば、巨大なベクターデータを持っていたとします。地物の数が膨大にあるような場合です(または複雑)。このような際には、大量のデータを扱うため計算や描画に時間を要することがあります。
筆ポリゴンが良い例でしょう。田畑の大きさと位置を持った筆ポリゴンは非常に細かいデータの集合で描画や計算に使うには向かない場合が多いです。そのため今回は、上で作成したポリゴンの範囲内にのみ含まれる筆ポリゴンを対象にしてみます。
つまりベースとなるポリゴンの内部に含まれている田畑のみを抽出するということです。
一方で大きな地物で構成されるような場合には、その形状が複雑さ抑える方法で扱いやすくすることができます。
他にはベクターをラスターにしてしまい(ラスタライズ)、それを利用して対象となるラスター画像をくり抜くという方法もあります(基本の考えは複雑なベクターを単純にするという意味で同じですが、この方法であれば大量の地物がある場合でも対処できます。しかしデータとしては荒くなるので、そこらは精度とのトレードオフだと思います)。
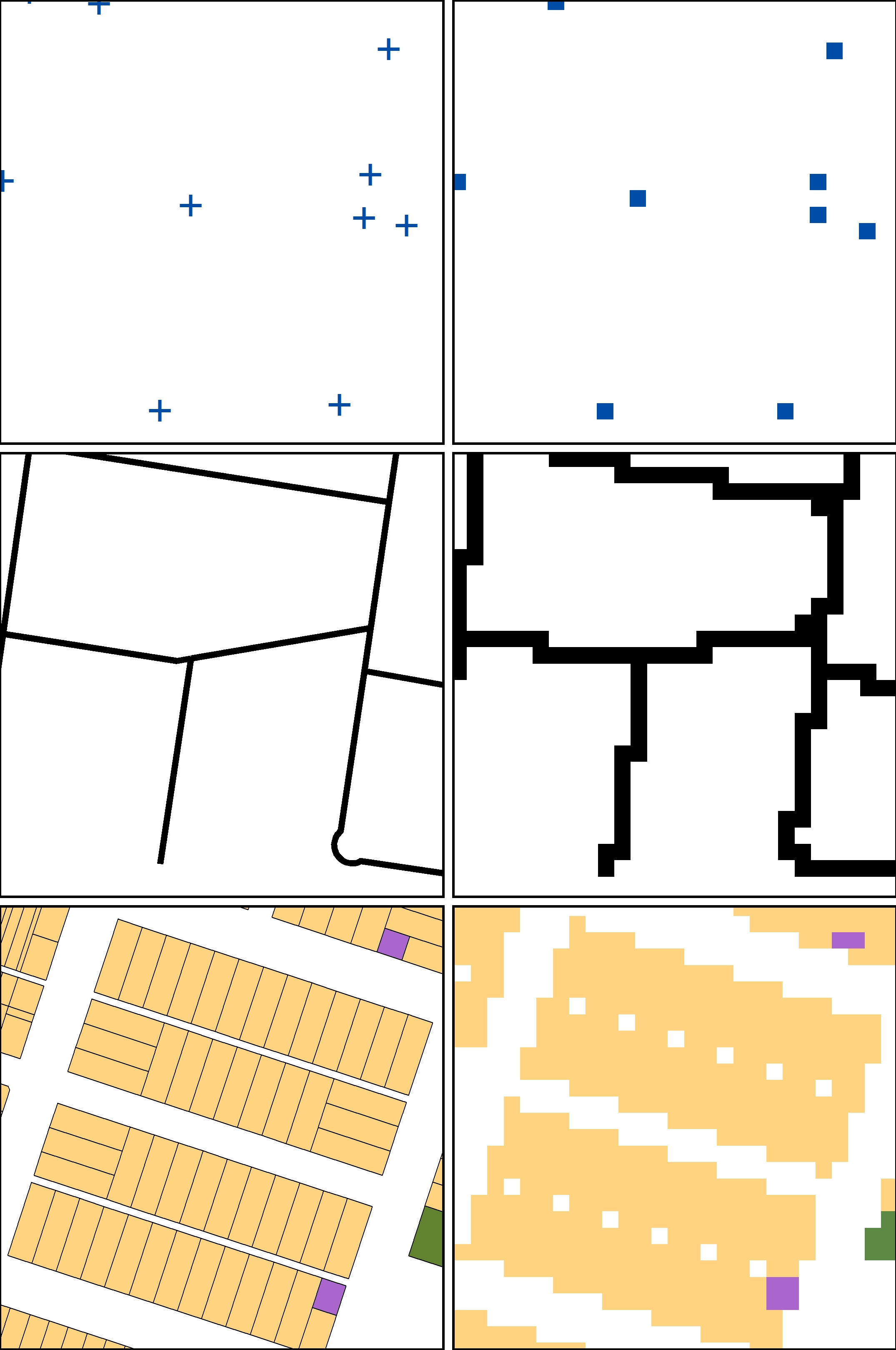
下図では、ラスターとベクターで表現されるものを対比しています。一番下の図が上の説明に当たる部分になります。複数のベクターで描かれた地物が、大きなラスター画像で単純に示されているのがわかると思います。
山形県鶴岡市の筆ポリゴンを取得し、そのGeoJSONをGeoPandasで読み込みます。
筆ポリゴンはこちらからダウンロード可能です。
# ファイルパスは適宜変更ください
fudeShounai = gpd.read_file('/content/drive/MyDrive/Sorabatake/062032019_5.geojson')
fudeShounai.head()
同様にCRSを確かめます。
fudeShounai.crsName: WGS 84
Axis Info [ellipsoidal]:
– Lat[north]: Geodetic latitude (degree)
– Lon[east]: Geodetic longitude (degree)
Area of Use:
– name: World.
– bounds: (-180.0, -90.0, 180.0, 90.0)
Datum: World Geodetic System 1984 ensemble
– Ellipsoid: WGS 84
– Prime Meridian: Greenwich
耕地の種類から、畑のみを抽出します。
fudeShounaiF = fudeShounai.loc[fudeShounai['耕地の種類'] == '畑',:]fudeShounaiF.plot(figsize=(12,12))
plt.show()
座標系を変換し、こちらも面積を求めてみましょう。# Convert 4326 to 6691
fudeShounaiF.to_crs('epsg:6691',inplace=True)
fudeShounaiF.insert(1,'area_ha',fudeShounaiF.geometry.area/10**4)fudeShounaiF.head()
shounaiのベクター内に含まれている筆ポリゴンのみを抽出します。
# Reference about .overlay: https://atmamani.github.io/cheatsheets/open-geo/geopandas-3/
mergedGdf = gpd.overlay(shounai,fudeShounaiF,how='intersection')
mergedGdf['areaIntersect'] = mergedGdf['geometry'].area/10**4mergedGdf.plot(figsize=(12,12))
先ほど描画した結果と異なり、北西の一部が切り取られていることが分かります。
foliumで確かめてみます。
latlon = [38.7918,139.8118]
m = folium.Map(latlon,zoom_start=14, control_scale=True)
folium.GeoJson(mergedGdf).add_to(m)
m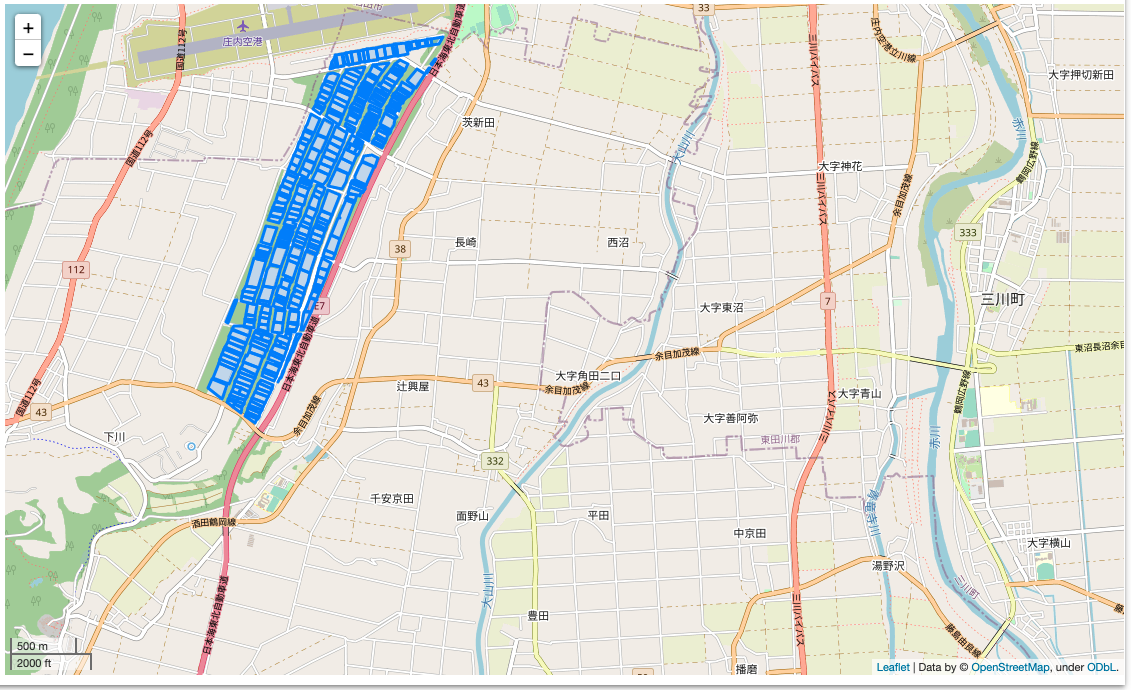
mergedGdf.head()
mergedGdf.areaIntersect.sum() # 合計は約108ha実行すると、108.00578337861548と出力されます。
結合したファイルをshpとして保存します。これは次の作業で利用します。
# ファイルへのパスは適宜変更してください
mergedGdf.drop(columns={'area_ha_1','耕地の種類','area_ha_2'},inplace=True) # 余計な列の削除
mergedGdf.to_file("/content/drive/MyDrive/Sorabatake/merged/mergedShounai.shp")衛星データの取得
今回はGoogle Earth Engineから衛星データを取得します。
取得するデータはMODIS/Terra Vegetation Index(MOD13Q1) Version 6です。
-
・16日周期で得られるデータです
-
・地上分解能は250mとなります
GEEを使う前に認証を行う必要があります。認証はGEEに登録済みのユーザーアカウントで行ってください。
import ee
ee.Authenticate()
ee.Initialize()Successfully saved authorization token.
と出れば認証に成功です。
先ほど保存したmegedShounai.shpをGEEにインポートします。
geemapを利用することにより、簡単にローカル(またはgoogle drive上)のファイルをGEEにインポートすることが可能です。
インポートした結果も簡単に表示をすることができます。
やや脇道にそれますが、geemapはeeの高機能なAPIを簡単にpython上で実行できるようにしたライブラリです。開発者であるWu博士は、非常に開発頻度が高く、また利用方法に関してのドキュメントも豊富に用意してありますので、是非ご覧ください。
Map = geemap.Map(center=latlon, zoom=14, control_scale=True)
fudePoly = geemap.shp_to_ee('/content/drive/MyDrive/Sorabatake/merged/mergedShounai.shp')
# fudePolySimple = fudePoly.map(lambda feature: feature.simplify(maxError = 50))
Map.addLayer(fudePoly, {}, 'Shounai Hatake') # レイヤー名はShounai Hatake
Map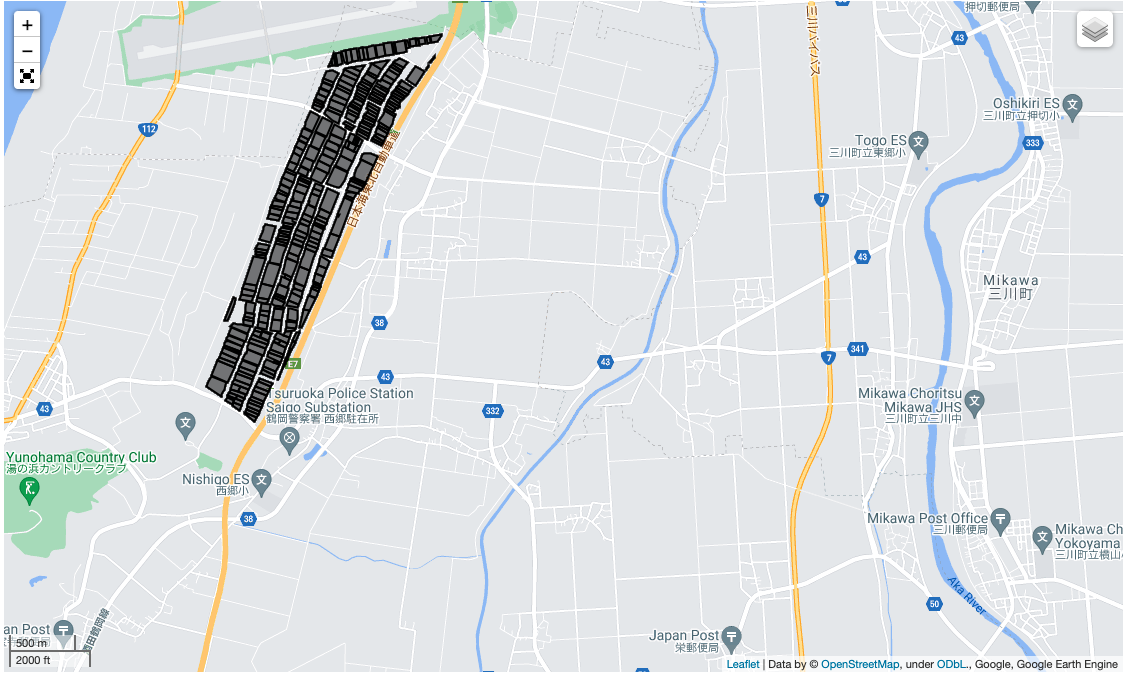
MOD13Q1からデータを取得します。Earth Engineでは、ラスターはImageCollection、ベクターはFeatureCollectionというコンストラクタ(メソッド)を用いて取得します。その名前の通り、取得するデータは全て大きなデータのまとまりになっています。
つまり、ee.ImageCollection(…)のようにすると、指定したデータでEarth Engineが持っている全てのデータ(アセット)を読み込むことになります。そのため、その後にデータを絞る必要があります。そこで用いられるのが、filterDateやfilterBoundsなどです。
# MODISデータの読み込み
# 今回は4月から9月までの範囲でデータを取得しています。
modis = ee.ImageCollection('MODIS/006/MOD13Q1') # IDの指定。今回はMODIS/006/MOD13Q1
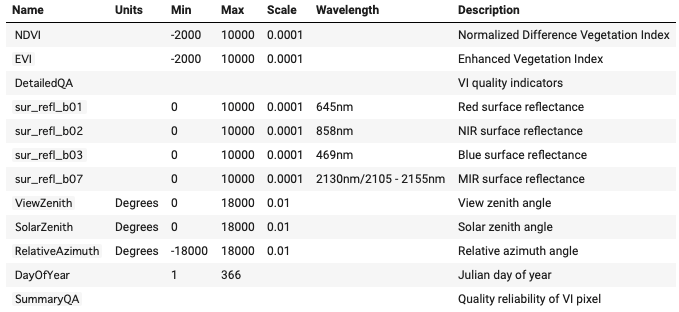
modis = modis.filterDate(ee.DateRange('2019-04-01','2019-7-01')) # 2019年4月1日から2019年7月1日を取得MOD13Q1に含まれるバンド情報を確認します。
modis.first().bandNames().getInfo()[‘NDVI’,
‘EVI’,
‘DetailedQA’,
‘sur_refl_b01’,
‘sur_refl_b02’,
‘sur_refl_b03’,
‘sur_refl_b07’,
‘ViewZenith’,
‘SolarZenith’,
‘RelativeAzimuth’,
‘DayOfYear’,
‘SummaryQA’]
# 今回はNDVIのみ取得
# evi = modis.select('EVI')
ndvi = modis.select('NDVI')NDVIのみを抽出しましょう。
スケーリングが必要なため関数を定義し、値を調整します。
詳しくは公式のドキュメント、Layersを開いてください。Scale Factorと書かれているところが利用する値となります。
# スケーリングのための関数
def scale_factor(image):
return image.multiply(0.0001).copyProperties(image, ['system:time_start'])
# 取得した各々のデータに対して上の関数を実行(Mapping)
# scaled_evi = evi.map(scale_factor)
scaled_ndvi = ndvi.map(scale_factor)gmap = geemap.Map(center=latlon, zoom=14, control_scale=True)
gmap.addLayer(scaled_ndvi.mean().clip(fudePoly), name='Averaged NDVI',# 4月から9月までの平均値を描画。clipでfudePoly内のピクセルだけ取得
vis_params={'min': 0,
'max': 1,
'palette': ['red', 'yellow','green']})
gmap.addLayerControl()
gmap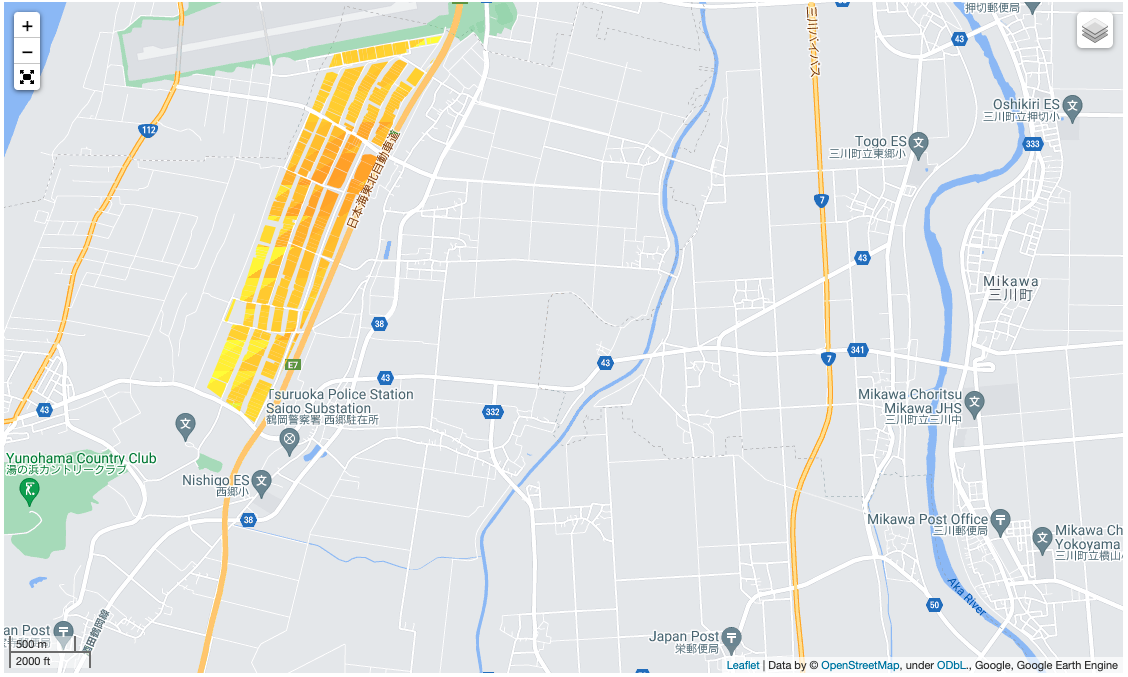
色が濃い部分がより緑が繁茂している状態にあると考えて良いです。同じ場所の畑ですが、値にわずかな違いがみられるのが面白いですね。
グラフも描画しましょう。こちらはipygeeを利用しています。
ipygeeではグラフを簡単に作成できるだけでなく、そのままデータフレームにできるので大変便利です。
(仮にipygeeが動かないという場合は、ここの部分を飛ばしても大丈夫です。データフレーム化もeeから出力されるデータを用いて作成することが可能です。)
# Shounai: MOD13Q1 EVI time series
shounai_ndvi = chart.Image.series(**{'imageCollection': scaled_ndvi,
'region': fudePoly,
'reducer': ee.Reducer.mean(),
'scale': 250, # 250m
'xProperty': 'system:time_start'})
shounai_ndvi.renderWidget(width='70%')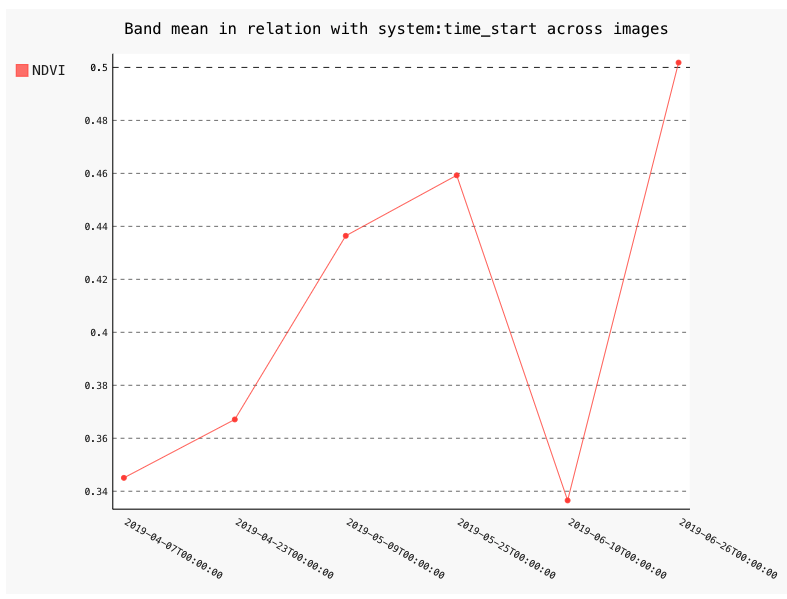
グラフは簡易的なものなので、実際にはmatplotlibなどを利用するのが良いです。上記で得れらたデータは簡単にテーブルデータに変更でき、この点が使いやすいと思います。
ndviDf = shounai_ndvi.dataframe
ndviDf
念のために、上の方法を利用しないテーブルデータの作成方法についても言及をします。
関心領域(この場合はfudePoly)からNDVIの平均値を取得し、その日付と平均値を取得するための関数を作成します。
# 関心領域のピクセルから平均値を導出
def aoi_mean(img):
# scaleには通常、そのラスターの解像度を渡します
# mean = img.reduceRegion(reducer=ee.Reducer.mean().unweighted(), geometry=fudePoly, scale=250).get('NDVI') # 重みなし
# mean = img.reduceRegion(reducer=ee.Reducer.mean(), geometry=fudePoly).get('NDVI') # 重みなし
mean = img.reduceRegion(reducer=ee.Reducer.mean(), geometry=fudePoly, scale=250).get('NDVI') # 重みあり
return img.set('date', img.date().format('Y-M-d')).set('mean',mean)reduceRegionでは、対象とする地域を絞りつつ、reducerによる重み付け平均を行っています。
ee.Reducerには他にも様々な種類があります。

上記で得られたee.Image(各々の画像データ)に対して、メタデータを付与しています。この場合は、日付(date)と計算した平均値(mean)です。
先ほども登場したmapですが、Earth Engineでは基本的にfor文を使うことはありません。ImageCollectionは画像データの集まりなので、気持ちとしてはこれらのデータそれぞれにfor文で上の処理を行いたいところです。しかし、Earth Engineではfor文は推奨されていません。その理由として、高度な並列処理ができなくなるからだそうです。
詳しくは、公式の情報をご覧ください。
ndvi_reduced_imgs = scaled_ndvi.map(aoi_mean)nested_list = ndvi_reduced_imgs.reduceColumns(ee.Reducer.toList(2), ['date','mean']).values().get(0)平均値を求めたImageCollectionから、指定した列のみを抽出したい場合、reduceColumnsを利用します。
reduceColumnsでは出力は辞書型になりますので、ee.Reducer.toListをを用いて出力をリスト型に変えています。最後に値のみを抽出し、リストの内側のみの値を取り出します。
これでデータフレームを作成する準備ができました。
df = pd.DataFrame(nested_list.getInfo(), columns=['date','ndvi'])
df['date'] = pd.to_datetime(df.date, format='%Y-%m-%d')df
描画を行います。
plt.rc('font', size=12)
fig, ax = plt.subplots(figsize=(12, 6))
# 線の見た目調整
ax.plot(df.date, df.ndvi, color='tab:orange', label='NDVI')
# ラベルとタイトルなど
ax.set_xlabel('')
ax.set_ylabel('NDVI')
ax.set_title('NDVI in a Shounai area')
ax.grid(True)
ax.legend(loc='upper left'); # レジェンドの位置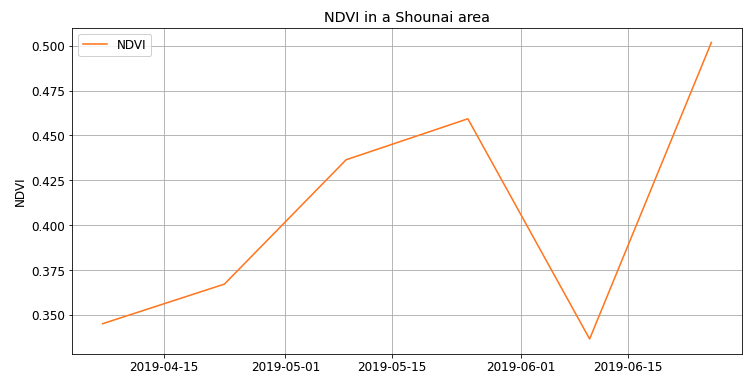
データフレーム化できました。
やや面倒ですが、このような作業で自分の興味範囲のみの衛星画像データをデータフレームにすることができます。今回は筆ポリゴンにより対象区画の面積も求めていますので、取得したNDVI(またはEVI)やその他のデータ(例えば気温などの気象データ)を組み合わせて収量予測のモデルが作れれば、面積あたりの収量を予測することも可能です。
筆ポリゴンの良さは、正確に農地の区画を区切ることが可能な点だと思います。区画は200m四方の精度になりますので、250m解像度のMODISデータとも相性が良いです。中山間における圃場の一つ一つというのは流石に難しいですが、我が町(村)の収穫量という意味では十分に利用可能かと考えます。
今回は農地の筆ポリゴンの話でしたが、近年、森林の情報も公開されています。自治体が提供する民有林の情報は「森林簿」として、森林の所在地、所有者、面積、樹種などのデータを有したものになります。これらのデータとNDVIなどを組み合わせれば、その町の樹林の健康度や樹林の広がりや縮少・分断化が分かりますし、季節性を考慮すれば、おおよそ落葉樹と常緑樹の構成比が分かるはずです。
様々なデータを探して、どれが衛星データと相性が良さそうか考えながら解析を行うのも楽しいと思います。ぜひ、色々と試してみてください。
補足
別の方法でも見ていきましょう。
まずはMOD13Q1に含まれる情報のおさらいです。
# 興味範囲を抽出。全てのバンドデータを取得する
aoiInfo = modis.getRegion(fudePoly,scale = 250).getInfo()allBandDf = pd.DataFrame(aoiInfo) # データフレーム作成
headers = allBandDf.iloc[0] # ヘッダ情報の取得(列名)
modisDf = pd.DataFrame(allBandDf.values[1:], columns=headers)
modisDf.head()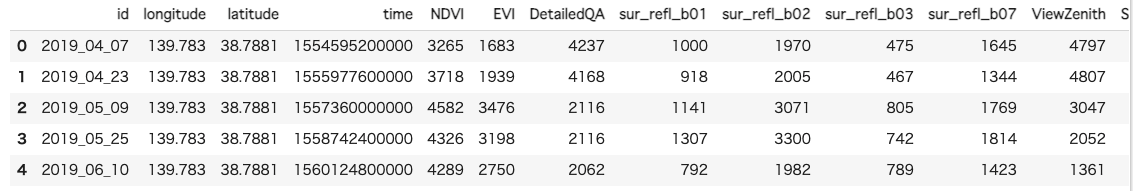
このように対象領域の全てのバンドデータを取得することも可能です。
下のように関数を定義すれば、より簡単に任意のバンドデータからデータフレームを作成することができます。
# Reference: https://developers.google.com/earth-engine/tutorials/community/intro-to-python-api-guiattard
# list_of_bandsにはバンド(波長)のデータを入れる
def ee_array_to_df(arr, list_of_bands):
"""ee.Image.getRegion array to pandas.DataFrameへ変換"""
df = pd.DataFrame(arr)
# ヘッダ追加
headers = df.iloc[0]
df = pd.DataFrame(df.values[1:], columns=headers)
# 緯度経度とtime、さらに選択したバンド情報を選択。加えて欠損値の削除する
df = df[['longitude', 'latitude', 'time', *list_of_bands]].dropna()
# バンドデータをnumericへ
for band in list_of_bands:
df[band] = pd.to_numeric(df[band], errors='coerce')
# timeをdatetime型に変更
df['datetime'] = pd.to_datetime(df['time'], unit='ms')
# 対象のバンド情報とtime, datetimeの列のみを残す
df = df[['time','datetime', *list_of_bands]]
return dflst_df_modis = ee_array_to_df(aoiInfo,['NDVI','EVI'])
lst_df_modis.NDVI = lst_df_modis.NDVI.multiply(0.0001)
lst_df_modis.EVI = lst_df_modis.EVI.multiply(0.0001)重みなし平均の結果は以下の通りです。
lst_df_modis.groupby('datetime').mean()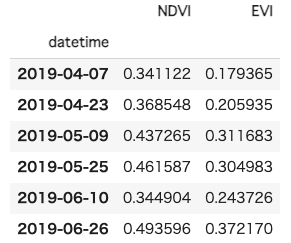
modisDfをGeoDataFrameに変更しましょう。
modisGdf = gpd.GeoDataFrame(
modisDf, geometry=gpd.points_from_xy(modisDf.longitude, modisDf.latitude),crs={'init': 'epsg:4326'})
# 日付の列を追加
modisGdf['date'] = pd.to_datetime(modisGdf.id,format='%Y_%m_%d')
ndviGdf = modisGdf.loc[:,['date','NDVI','geometry']].copy()
ndviGdf['scaledNdvi'] = ndviGdf.NDVI.astype('int64').multiply(0.0001)ndviGdf.head()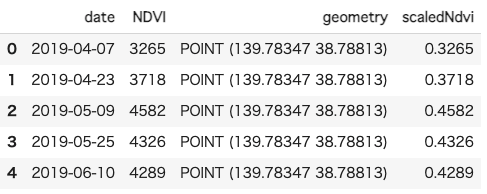
ndviGdf.geometry.unique()ndviGdf.plot(figsize=(6,12))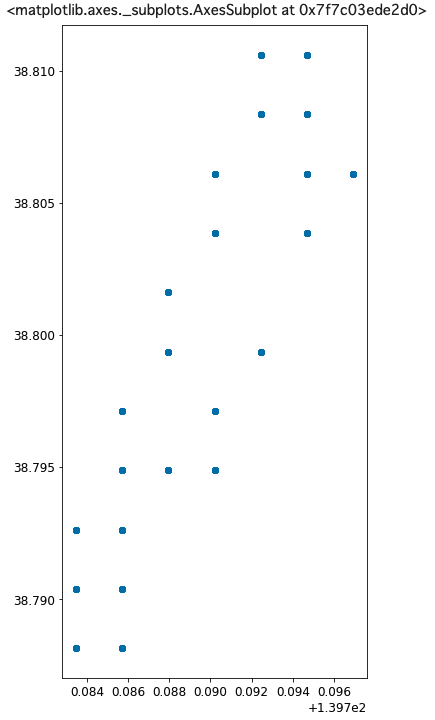
getRegionでscaleを250mにした結果、関心領域から23点が選ばれていることがわかります。fudePolyはmergedGdfから作成したので、302個の図形(地物)から成っています。
実はここらの処理はあまりよくわかっていないのですがscaleにはかなり揺らぎがあり、基本的に画像データの分解能より余程小さくしないかぎり、参照点の数は増えません。
関心領域全体の平均を計算するという点において、ここの数は平均に影響をしてきません、あまり神経質にならなくてよいかと思います(重みを考慮すると変わります)。
またポリゴン一つ一つの値を見たいという場合にはさらに複雑な処理が必要になります(ここでは触れませんが)。
参照系を変更し、どの位置に参照点があるのかを確かめます。
ndviGdf = ndviGdf.to_crs('epsg:6691')ポイントデータになっていますので(reducerを使ってまとめたため)、ポイントからポリゴン(四角形)を作成しましょう。
from shapely.geometry import Point
# Buffer the points using a square cap style
# Note cap_style: round = 1, flat = 100, square = 3
ndviGdf['buffer'] = ndviGdf.geometry.buffer(100, cap_style = 3) # 100m buffer描画してみます。
ndviGdf.loc[:,'buffer'].plot(figsize=(6,12),edgecolor='k')
eeはいろいろと深く掘らないと、わからないことも多いです。
上のような処理で問題がある場合には、くり抜いた画像をtiffで出力し、その画像に対してはrasterstatsなどでゾーン統計をとるという方法もあります(関心領域内の平均や和などは変わりません)。
是非いろいろと遊んでみてください。
次回はゾーン統計について、さらに深く学んでいきましょう。
以上でGeoPandas入門(後編)も終わります。
今回はPythonでの紹介でしたので、ee+geemapを中心とした紹介になりました。
RでEEを利用する場合には、rgeeがあります。こちらもかなり便利になっていますので、是非活用してください。
参考文献
MODIS Vegetation Indices: a GEE Approach
GeoSpatial analysis in Python and Jupyter Notebooks
Automating GIS and remote sensing workflows with open python libraries


