植物の分布から田畑の利用状況を推測する衛星データの利活用方法
Tellusでは、利便性(データのCOG化)と検索精度(APIのフォーマット統一)の向上を目指して、データ配信用APIの更新を行いました。それに伴い、今回は新しい配信用APIを利用してAVNIR-2のデータを取得する方法について、田畑の利用状況を推定する例を通してご紹介します。
Tellusでは、利便性(データのCOG化)と検索精度(APIのフォーマット統一)の向上を目指して、データ配信用APIの更新を行いました。それに伴い、今回は新しい配信用APIを利用してAVNIR-2のデータを取得する方法についてご紹介します。
https://www.tellusxdp.com/ja/news/20220630_000502.html
Tellusの解析環境を契約していない方であっても利用可能なAPIを用いて解析を行いますので、皆さまお手元の環境でお試しください。
なお、本記事は技術評論社から出版されている月刊誌Software Designに宙畑が寄稿した連載「衛星データプラットフォームTellusハンズオン」の内容から、一部修正して掲載するものです。
衛星データで分かること・できること
宙畑では過去にも何度か紹介していますが、改めて「波長」とは文字通り波の長さのことです。色や通信に使う電波や赤外線、紫外線などすべて電磁波であり、それぞれ「波長」によって異なる性質を持ちます。
詳しくはこちらをご覧ください:
光の波長って何? なぜ人工衛星は人間の目に見えないものが見えるのか
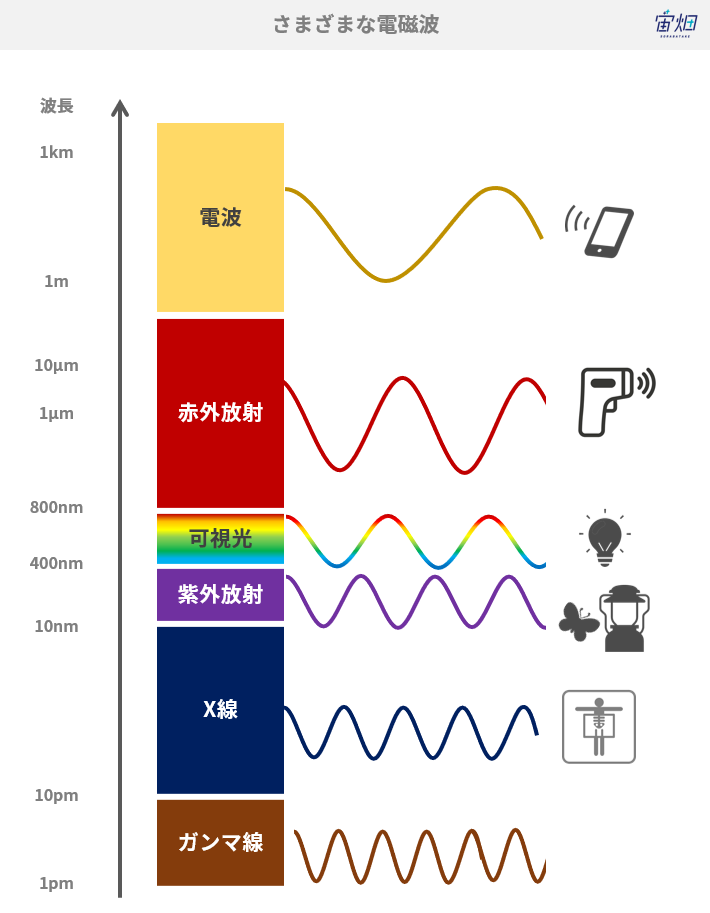
人の目はこの電磁波の中で可視光といわれる限られた範囲の波長帯しか認識(観測)できません。この可視光の波長帯を青、緑、赤の色の組み合わせで捉えています。
これに対して、人工衛星のセンサーでは可視光以外の波長帯も観測できます。観測している波長の中には、紫外線や赤外線、電波をとらえるセンサーを搭載しているものもあるので、私たちが目にする地球とは異なった視点で地球の姿を見ることができます。
波長によってなぜ見え方が違うのでしょうか。それは対象によって吸収(透過)あるいは反射する太陽光の波長が異なるからです。
例えば、植物は他の物質に比べて近赤外線※の帯域で強く反射することが分かっています。つまり、近赤外線で観測した結果を利用すると、植物の分布を調べやすいと言えます。
※近赤外線(きんせきがいせん):可視光に近い波長の赤外線
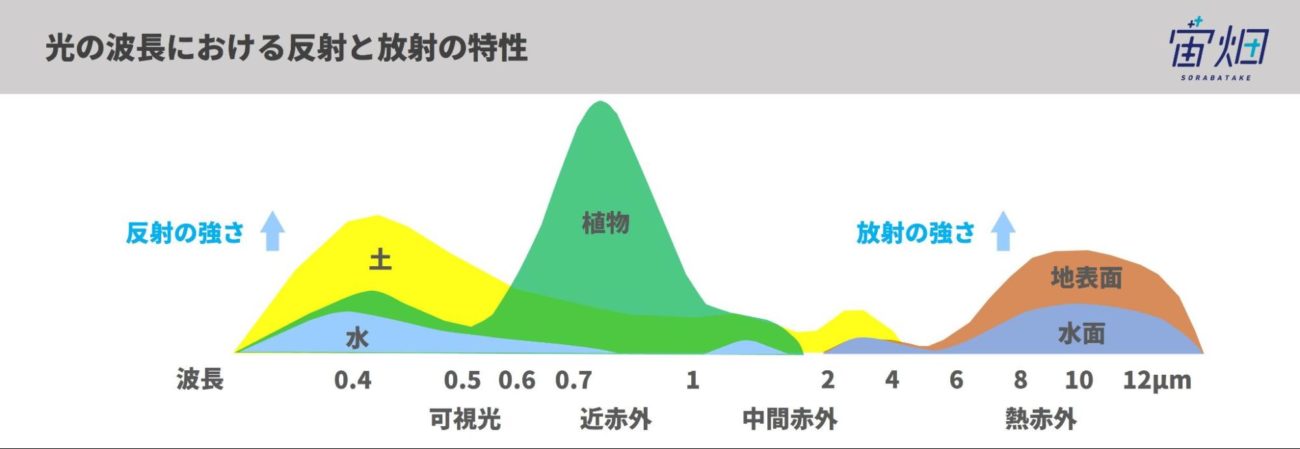
さらに、それぞれ個別の波長で調べるだけではなく、複数の波長のデータを組み合わせて計算することで調べたいものが判別しやすくなる場合があります。
今回は、よく利用される波長の組み合わせである正規化植生指数(NDVI)という指数について解析事例を交えて紹介します。
正規化植生指数=NDVI(Normalized Difference Vegetation Index)とは、植物の活性度を調べる指数のことです。近赤外線の光の強さ(NIR)と赤色の光の強さ(Red)を以下の式に当てはめることで求めることができます。
NDVI=(NIR-Red)/(NIR+Red)
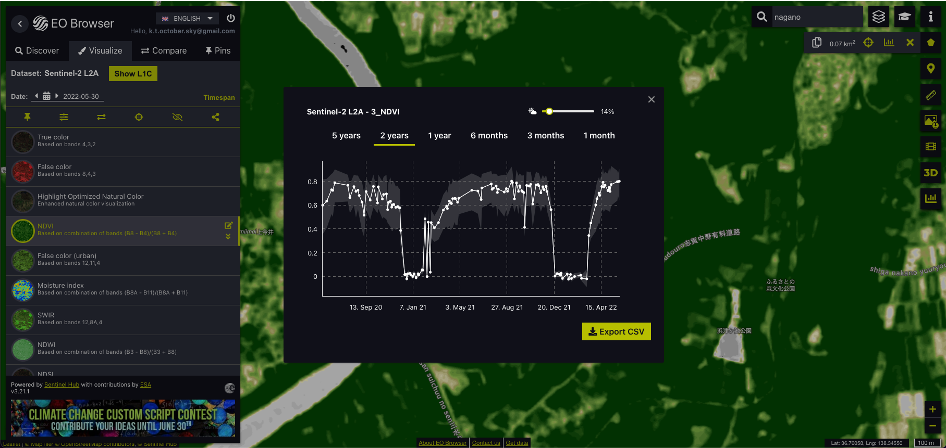
例えば、以下のように過去何年か分のNDVIの値を時系列に見ていくことで、農作物の生育状況を調べることができます。
衛星データプラットフォームTellus登録と準備
TellusのAPIを利用するためには、Tellusのアカウント登録が必要です。まだアカウントをお持ちではない方は、以下のページで必要情報を入力し、アカウントを作成します。
https://www.tellusxdp.com/market/login/
Tellusで公開しているデータの利用規則はそれぞれのデータの提供元のデータポリシーに基づきます。中には、Tellusが提供する開発環境内での利用に限定され、ローカル環境へのダウンロードが禁止されているデータもあるので注意が必要です。
今回は、Tellus環境外からも利用可能なデータを使用しますので、Tellusアカウント登録をした上でAPIキーの発行さえすれば、お手元のJupyter NotebookやGoogle Colabなどでもお試しいただけます。
本記事ではGoogle Colabを例に紹介します。
まずは自分のGoogle Driveと接続します。
## google driveと接続
from google.colab import drive
drive.mount('/content/drive')解析に必要なライブラリをインポートします。
!pip install rasterio
!pip install geopandasimport os
import requests
import json
import glob
import rasterio.mask
import fiona
import numpy as np
import rasterio as rio
import matplotlib.pyplot as plt
import geopandas as gpd
from rasterio.plot import show
from rasterio.plot import show_histAPI利用の際に必要なトークンは、Tellusにログイン後、右上のユーザー名のプルダウンメニューから[開発環境]>[APIアクセス]を選択し、[トークンの発行]から新しいトークンを発行します。
#各個人のAPIトークンを入力
token = 'XXXXXXXXXXXXXXXX'衛星データの検索・取得
Tellusの衛星データAPIのリファレンスは以下で公開しています。
https://www.tellusxdp.com/docs/travelers/
Tellusで公開している衛星データは「データセット」「シーン」「ファイル」という構造になっています。
▼Tellusのデータ構造
データセットA
└ シーンa
└ ファイルa-i
└ ファイルa-ii
└ シーンb
└ ファイルb-i
└ ファイルb-i
・・・
「データセット」はある衛星に搭載されたセンサや、前処理方式などが揃ったデータのまとまりです。
「シーン」は特定の場所をあるタイミングで撮影したまとまりで、撮影の方法などにより、例えば赤青緑の波長の要素毎に複数の「ファイル」が存在します。
詳しくはこちらの記事等をご覧ください。
TellusのCOG形式のAPIを使って、ASNARO-2の画像を重ね合わせて前後の変化抽出を実施してみた
衛星データをAPIで取得する前に、Tellus Traveler(ブラウザ)上で必要な衛星データに関する情報を取得します。Travelerの使い方は以下をご覧ください。
https://www.tellusxdp.com/ja/traveler-service/guide/traveler-how-to/2_traveler.html
今回は、2006年10月31日にAVNIR-2によって撮影された青森県の画像を利用します。ご自身で興味のある領域を検索し、データセットIDとシーンIDを取得して解析にチャレンジしてみてください。
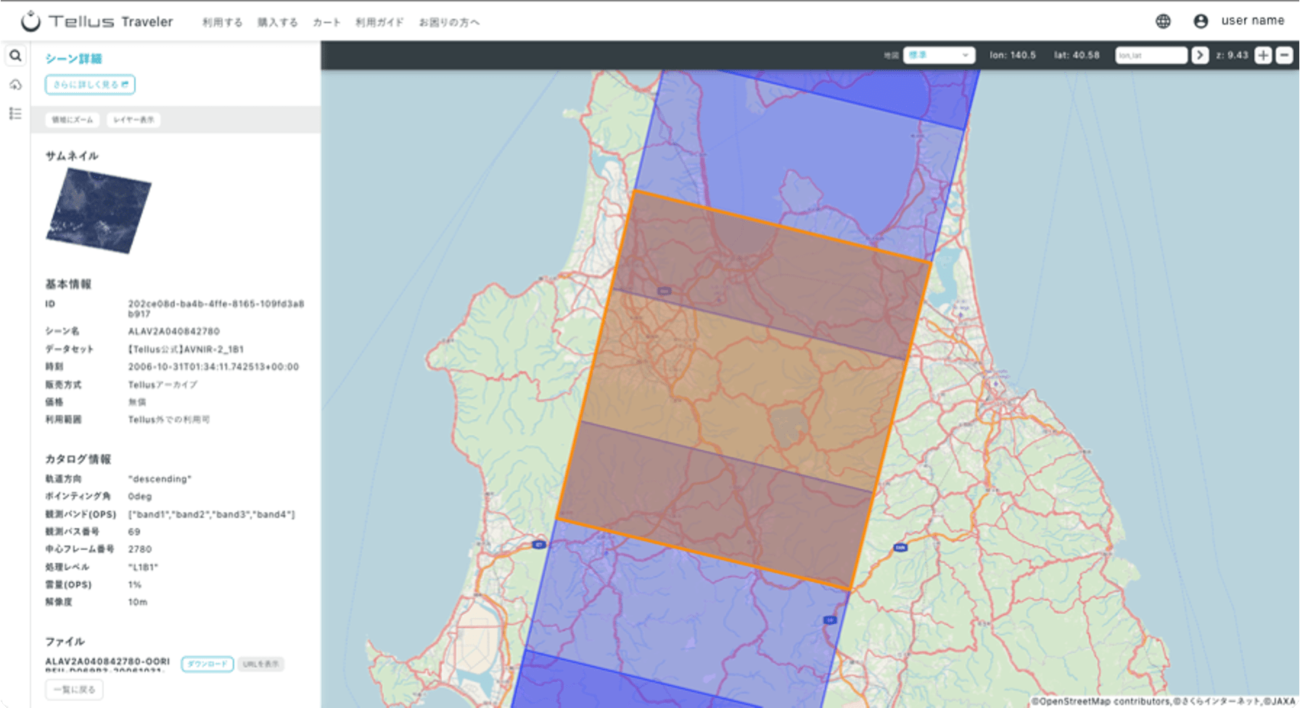
上記の衛星画像のデータセットIDとシーンIDは以下の通りです。
dataset_id='ea71ef6e-9569-49fc-be16-ba98d876fb73'
scenes = [
'202ce08d-ba4b-4ffe-8165-109fd3a8b917'
]調べたデータセットIDとシーンIDから、そのシーンに含まれるファイル名を取得します。Tellus Travelerを利用してデータを取得する関数は以下の通りです。
# データセットIDとシーンIDから、そのシーンに含まれるファイル名を取得する
def get_dataset_data_by_id_files(base_path, token, dataset_id, data_id):
print(base_path, token, dataset_id, data_id)
url = '{}/api/traveler/v1/datasets/{}/data/{}/files/'.format(
base_path, dataset_id, data_id)
headers = {
'Authorization': 'Bearer ' + token,
'content-type': 'application/json'
}
r = requests.get(url, headers=headers)
assert r.status_code == 200
return json.loads(r.content)次に、ファイルIDを指定してデータのダウンロードURLを取得します。
# ファイルIDを指定して、ダウンロードURLを取得する
def get_dataset_data_by_id_files_by_id_download_url(base_path, token, dataset_id, data_id, file_id):
url = '{}/api/traveler/v1/datasets/{}/data/{}/files/{}/download-url/'.format(
base_path, dataset_id, data_id, file_id)
headers = {
'Authorization': 'Bearer ' + token,
'content-type': 'application/json'
}
r = requests.post(url, headers=headers)
assert r.status_code == 200
return json.loads(r.content)ダウンロードURLを用いて衛星データをダウンロードする関数は以下の通りです。
# 衛星データのダウンロードを行う
def download(scenes, dataset_id, token, dist='./', base_path='https://www.tellusxdp.com'):
for scene_id in scenes:
files = get_dataset_data_by_id_files(
base_path, token, dataset_id, scene_id)
print(files)
rawdata = files['results']
path = os.path.join(dist, scene_id)
if len(rawdata) > 0:
for file in rawdata:
file_id = file['id']
file_name = file['name']
file_path = os.path.join(path, file_name)
download_url = get_dataset_data_by_id_files_by_id_download_url(
base_path, token, dataset_id, scene_id, file_id)['download_url']
r = requests.get(download_url, stream=True)
if not os.path.exists(path):
os.makedirs(path)
with open(file_path, 'wb') as f:
for chunk in r.iter_content(chunk_size=1024):
if chunk:
f.write(chunk)
f.flush()これらの関数を実行することで、衛星データをご自身のGoogle Driveにダウンロードできます。
# データDL
download(scenes, dataset_id, token)NDVIの算出
さて、様々なバンドのデータがダウンロードできたらそのデータを処理してNDVIを算出していきましょう。
本来NDVIは輝度値ではなく、輝度値に大気補正処理を行い算出する地上反射率を利用しますが、今回は処理が簡素な輝度値に変換して抽出しています。
# NDVIの算出
def mk_ndvi_rio(red_img, nir_img, red_gain, red_offset, nir_gain, nir_offset, out_ndvi):
red = rio.open(red_img)
nir = rio.open(nir_img)
red_arr = red.read().astype(float)
nir_arr = nir.read().astype(float)
kwargs = {
"width" : red.width,
"height" : red.height,
"count" : 1,
"crs" : red.crs,
"transform" : red.transform,
"dtype" : rio.float32}
## DN2輝度変換
red_arr = red_arr * red_gain + red_offset
nir_arr = nir_arr * nir_gain + nir_offset
## NDVI算出
ndvi = np.where(((red_arr != 0) & (nir_arr != 0)),((nir_arr - red_arr) / (nir_arr + red_arr) ),0)
write_ndvi = rio.open(out_ndvi,"w",driver="Gtiff",**kwargs)
write_ndvi.write(ndvi)
write_ndvi.close()
return(out_ndvi)NDVI値の画像を作成していきます。
DATA_DIR = "/content/drive/MyDrive/work/study/Sorabatake/02_data"
DIR_LIST = os.listdir(DATA_DIR)
NDVI_DIR = "/content/drive/MyDrive/work/study/Sorabatake/03_ndvi"
#print(DIR_LIST)
#データを取得
for dir_name in DIR_LIST:
dir_path = os.path.join(DATA_DIR, dir_name)
#print(dir_path)
subdir_list = os.listdir(dir_path)
red_path = glob.glob(dir_path + "/IMG-03-*.tif")
nir_path = glob.glob(dir_path + "/IMG-04-*.tif")
hdr_path = glob.glob(dir_path + "/HDR-*.txt")
hdr_path = str(hdr_path[0])
# HDR情報から、ゲイン、オフセット情報を抽出
hdr_f = open(hdr_path,"r")
hdr_data = hdr_f.read()
hdr_f.close
# gain, offset
red_gain = float(hdr_data[1753:1761])
red_offset = float(hdr_data[1761:1769])
nir_gain = float(hdr_data[1769:1777])
nir_offset = float(hdr_data[1777:1785])
print(red_gain, red_offset, nir_gain, nir_offset)
red_path = red_path[0]
nir_path = nir_path[0]
print(red_path, nir_path)
#NDVI画像の作成
base_name = os.path.basename(red_path)
ndvi_file = "NDVI-" + base_name[7:]
ndvi_path = os.path.join(NDVI_DIR, ndvi_file)作成したNDVI画像を表示します。
NDVI_20061031 = rio.open(NDVI_DIR + "/NDVI-ALAV2A040842780-OORIRFU-D069P3-20061031-001.tif")
NDVI_20061031 = NDVI_20061031.read(1)
fig = plt.figure(figsize=(18,18))
img1 = ax1.imshow(NDVI_20061031, cmap='RdYlGn', vmin=-1, vmax=1)
ax1.set_title("NDVI_20061031")
cbar1 = fig.colorbar(img1,ax=ax1, aspect=40,pad=0.08,shrink=0.8)
plt.show(fig)
以下のような画像が表示されます。
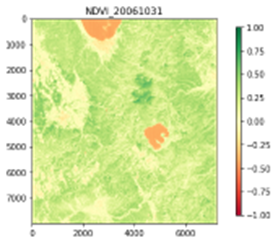 |
 |
植物が活発なところを濃い緑で、植物のないところ(湖など)が赤で表示されています。一般的なカラー画像(右)と比較しても、植物が活性化している部分がより分かりやすくなっています。
NDVIを時系列にグラフ化する
ここまでは、1枚の衛星データについて分析をしてきましたが、同じ場所の衛星データを時系列で見ていくことで、田畑の利用状況を推定してみます。例えば、夏場にNDVIが低いところがあれば、その田畑は作付けが行われていない、つまりは遊休農地である可能性があると考えることができます。
そこで、農林水産省が公開している筆ポリゴンと呼ばれる田畑の区画データを使って、区画ごとのNDVI値の時系列推移を求めてみましょう。
「衛星データの検索・取得」の節のコードを用いてデータをダウンロードします。ダウンロードしたデータからNDVIを算出していきます。「衛星データの検索・取得」の続きとして以下を実行してください。
def mk_ndvi_rio(red_img, nir_img, red_gain, red_offset, nir_gain, nir_offset, out_ndvi):
"""
NAME:
make_ndvi_rio
ARGUMENT:
red_img : 赤バンド画象
nir_img : 赤外パンド画像
red_gain : 赤バンドゲイン
red_offset: 赤バンドオフセット
nir_gain : 赤外バンドゲイン
nir_offset: 赤外バンドオフセット
out_ndvi : NDVI画像
DISCRIPTION:
The original code was written by N.Furuta
■DN値から輝度値への変換式
L = a×O+b
L:輝度(W/m2/sr/μm)
O:校正済みデジタル値(DN値)
a:絶対校正係数(ゲイン値)
b:オフセット値
※プロダクトフォーマット説明書 AVNIR-2編、表3.3-8 アンシラリ2(ラジオメトリック校正)レコード、p3-30 参照
■NDVI値の計算
(近赤外バンド - 赤バンド)/(近赤外バンド + 赤バンド)
※本来は輝度値をさらに地上反射率へ変換したものを利用
※本関数では簡単のため輝度値を用いて計算(簡易NDVI)
RETURN:
out_ndvi : NDVI画像(パス)
"""
red = rio.open(red_img)
nir = rio.open(nir_img)
red_arr = red.read().astype(float)
nir_arr = nir.read().astype(float)
kwargs = {
"width" : red.width,
"height" : red.height,
"count" : 1,
"crs" : red.crs,
"transform" : red.transform,
"dtype" : rio.float32}
## DN2輝度変換
red_arr = red_arr * red_gain + red_offset
nir_arr = nir_arr * nir_gain + nir_offset
## NDVI算出
ndvi = np.where(((red_arr != 0) & (nir_arr != 0)),((nir_arr - red_arr) / (nir_arr + red_arr) ),0)
write_ndvi = rio.open(out_ndvi,"w",driver="Gtiff",**kwargs)
write_ndvi.write(ndvi)
write_ndvi.close()
return(out_ndvi)NDVI画像を作成します。
"""
2.NDVI画像の作成
## HDR情報は公式ドキュメントを参照
https://www.eorc.jaxa.jp/ALOS/alos-ori/doc/AVNIR-2_ORI_format_jp.pdf
"""
# 衛星画像(AVNIR-2)を保存しているフォルダを指定
# 自身の環境に合わせて指定先は変更してください
DATA_DIR = "/content/drive/MyDrive/work/study/Sorabatake/02_data"
DIR_LIST = os.listdir(DATA_DIR)
# NDVI画像の保存先のフォルダを指定
NDVI_DIR = "/content/drive/MyDrive/work/study/Sorabatake/03_ndvi"
#print(DIR_LIST)
#データを取得
for dir_name in DIR_LIST:
dir_path = os.path.join(DATA_DIR, dir_name)
#print(dir_path)
subdir_list = os.listdir(dir_path)
red_path = glob.glob(dir_path + "/IMG-03-*.tif")
nir_path = glob.glob(dir_path + "/IMG-04-*.tif")
hdr_path = glob.glob(dir_path + "/HDR-*.txt")
hdr_path = str(hdr_path[0])
#print(hdr_path)
# HDR情報から、ゲイン、オフセット情報を抽出
hdr_f = open(hdr_path,"r")
hdr_data = hdr_f.read()
hdr_f.close
# gain, offset
red_gain = float(hdr_data[1753:1761])
red_offset = float(hdr_data[1761:1769])
nir_gain = float(hdr_data[1769:1777])
nir_offset = float(hdr_data[1777:1785])
print(red_gain, red_offset, nir_gain, nir_offset)
red_path = red_path[0]
nir_path = nir_path[0]
print(red_path, nir_path)
#NDVI画像の作成
base_name = os.path.basename(red_path)
ndvi_file = "NDVI-" + base_name[7:]
ndvi_path = os.path.join(NDVI_DIR, ndvi_file)まず、目視で変化を観察してみましょう。
以下のコードを実行することで、複数時期の青森県域のNDVI画像を表示します。青森以外のデータを取得している方は以下のファイル名を修正してください。
#NDVI画像の表示
NDVI_20061031 = rio.open(NDVI_DIR + "/NDVI-ALAV2A040842780-OORIRFU-D069P3-20061031-001.tif")
NDVI_20070618 = rio.open(NDVI_DIR + "/NDVI-ALAV2A074392780-OORIRFU-D069P3-20070618-002.tif")
NDVI_20080805 = rio.open(NDVI_DIR + "/NDVI-ALAV2A134782780-OORIRFU-D069P3-20080805-003.tif")
NDVI_20081105 = rio.open(NDVI_DIR + "/NDVI-ALAV2A148202780-OORIRFU-D069P3-20081105-000.tif")
NDVI_20090508 = rio.open(NDVI_DIR + "/NDVI-ALAV2A175042780-OORIRFU-D069P3-20090508-003.tif")
NDVI_20100626 = rio.open(NDVI_DIR + "/NDVI-ALAV2A235432780-OORIRFU-D069P3-20100626-002.tif")
NDVI_20100926 = rio.open(NDVI_DIR + "/NDVI-ALAV2A248852780-OORIRFU-D069P3-20100926-001.tif")
NDVI_20061031 = NDVI_20061031.read(1)
NDVI_20070618 = NDVI_20070618.read(1)
NDVI_20080805 = NDVI_20080805.read(1)
NDVI_20081105 = NDVI_20081105.read(1)
NDVI_20090508 = NDVI_20090508.read(1)
NDVI_20100626 = NDVI_20100626.read(1)
NDVI_20100926 = NDVI_20100926.read(1)
fig = plt.figure(figsize=(18,18))
ax1 = fig.add_subplot(331)
ax2 = fig.add_subplot(332)
ax3 = fig.add_subplot(333)
ax4 = fig.add_subplot(334)
ax5 = fig.add_subplot(335)
ax6 = fig.add_subplot(336)
ax7 = fig.add_subplot(337)
img1 = ax1.imshow(NDVI_20061031, cmap='RdYlGn', vmin=-1, vmax=1)
ax1.set_title("NDVI_20061031")
cbar1 = fig.colorbar(img1,ax=ax1, aspect=40,pad=0.08,shrink=0.8)
img2 = ax2.imshow(NDVI_20070618, cmap='RdYlGn', vmin=-1, vmax=1)
ax2.set_title("NDVI_20070618")
cbar2 = fig.colorbar(img2,ax=ax2, aspect=40,pad=0.08,shrink=0.8)
img3 = ax3.imshow(NDVI_20080805, cmap='RdYlGn', vmin=-1, vmax=1)
ax3.set_title("NDVI_20080805")
cbar3 = fig.colorbar(img3,ax=ax3, aspect=40,pad=0.08,shrink=0.8)
img4 = ax4.imshow(NDVI_20081105, cmap='RdYlGn', vmin=-1, vmax=1)
ax4.set_title("NDVI_20081105")
cbar4 = fig.colorbar(img4,ax=ax4, aspect=40,pad=0.08,shrink=0.8)
img5 = ax5.imshow(NDVI_20090508, cmap='RdYlGn', vmin=-1, vmax=1)
ax5.set_title("NDVI_20090508")
cbar5 = fig.colorbar(img5,ax=ax5, aspect=40,pad=0.08,shrink=0.8)
img6 = ax6.imshow(NDVI_20100626, cmap='RdYlGn', vmin=-1, vmax=1)
ax6.set_title("NDVI_20100626")
cbar6 = fig.colorbar(img6,ax=ax6, aspect=40,pad=0.08,shrink=0.8)
img7 = ax7.imshow(NDVI_20100926, cmap='RdYlGn', vmin=-1, vmax=1)
ax7.set_title("NDVI_20100926")
cbar7 = fig.colorbar(img7,ax=ax7, aspect=40,pad=0.08,shrink=0.8)
plt.show(fig)そうすると以下のような結果を得られるかと思います。

今回は例として、青森県藤崎町を対象にNDVI解析を行っていきます。そのために上記で算出したNDVI画像を藤崎町の区画に沿って切り出します。
※藤崎町の区画ポリゴンは国土地理院が公開している国土数値情報のうち、行政区域データを利用しています。
# 青森県全域の行政区域データの中から藤崎町だけのDVI値を切り抜く
# 藤崎町のポリゴンを抽出
# 行政区域データの保存先のフォルダを指定
# ※自身の環境に合わせて指定先は変更してください
AOMORI_PATH = "/content/drive/MyDrive/work/study/Sorabatake/04_polygon/Aomori_N03-20210101_02_GML/"
# そのうちshape形式のデータを指定
AOMORI_SHP = "N03-21_02_210101.shp"
df_aomori = gpd.read_file(os.path.join(AOMORI_PATH, AOMORI_SHP), encoding="shift-jis")
Fujisaki_machi = df_aomori[df_aomori["N03_004"].isin(["藤崎町"])]
Fujisaki_machi = Fujisaki_machi.drop(columns=["N03_002","N03_003"])
#投影変換
reprojest_Fujisaki_machi = Fujisaki_machi.to_crs({"init":"epsg:32654"})
reproject_out = os.path.join(AOMORI_PATH, "epsg_32654-"+ AOMORI_SHP)
#出力
reprojest_Fujisaki_machi.to_file(driver="ESRI Shapefile",filename=reproject_out)
#GeoDataFrameを描画
f,ax = plt.subplots(1, figsize=(6,6))
ax = Fujisaki_machi.plot(axes=ax)
plt.show同じ場所で解析を実行した場合、上記を実行すると以下のような結果を得られます。
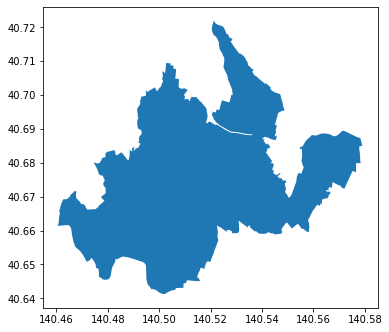
次に、NDVI画像を藤崎町のポリゴンでマスク処理し、藤崎市以外の範囲のNDVI画像を削除します。
# マスク処理済み画像の保存先のフォルダを指定
# ※自身の環境に合わせて指定先は変更してください
MASK_DIR = "/content/drive/MyDrive/work/study/Sorabatake/03_ndvi/02_fujisaki_masked"
ndvi_list = os.listdir(NDVI_DIR)
ndvi_list = [f for f in ndvi_list if os.path.isfile(os.path.join(NDVI_DIR, f))]
for ndvi_file_name in ndvi_list:
print("処理開始:" + ndvi_file_name)
with fiona.open(reproject_out, "r") as mask:
masks = [feature["geometry"] for feature in mask]
ndvi_data = os.path.join(NDVI_DIR, ndvi_file_name)
print(ndvi_data)
print(masks)
with rasterio.open(ndvi_data) as src:
out_image, out_transform = rasterio.mask.mask(src, masks, nodata=np.nan, crop=True)
out_meta = src.meta
out_meta.update({"driver": "GTiff",
"height": out_image.shape[1],
"width": out_image.shape[2],
"transform": out_transform})
with rasterio.open(MASK_DIR + "/Masked-" + str(ndvi_file_name), "w", **out_meta) as dst:
dst.write(out_image)次に、目視で確認するために藤崎町のNDVI画像を一覧で表示します。
#藤崎町で切り出したNDVI画像の表示
masked_NDVI_20061031 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A040842780-OORIRFU-D069P3-20061031-001.tif")
masked_NDVI_20070618 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A074392780-OORIRFU-D069P3-20070618-002.tif")
masked_NDVI_20080805 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A134782780-OORIRFU-D069P3-20080805-003.tif")
masked_NDVI_20081105 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A148202780-OORIRFU-D069P3-20081105-000.tif")
masked_NDVI_20090508 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A175042780-OORIRFU-D069P3-20090508-003.tif")
masked_NDVI_20100626 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A235432780-OORIRFU-D069P3-20100626-002.tif")
masked_NDVI_20100926 = rio.open(MASK_DIR + "/Masked-NDVI-ALAV2A248852780-OORIRFU-D069P3-20100926-001.tif")
masked_NDVI_20061031 = masked_NDVI_20061031.read(1)
masked_NDVI_20070618 = masked_NDVI_20070618.read(1)
masked_NDVI_20080805 = masked_NDVI_20080805.read(1)
masked_NDVI_20081105 = masked_NDVI_20081105.read(1)
masked_NDVI_20090508 = masked_NDVI_20090508.read(1)
masked_NDVI_20100626 = masked_NDVI_20100626.read(1)
masked_NDVI_20100926 = masked_NDVI_20100926.read(1)
fig = plt.figure(figsize=(16,12))
ax1 = fig.add_subplot(331)
ax2 = fig.add_subplot(332)
ax3 = fig.add_subplot(333)
ax4 = fig.add_subplot(334)
ax5 = fig.add_subplot(335)
ax6 = fig.add_subplot(336)
ax7 = fig.add_subplot(337)
img1 = ax1.imshow(masked_NDVI_20061031, cmap='RdYlGn', vmin=-1, vmax=1)
ax1.set_title("NDVI_20061031")
cbar1 = fig.colorbar(img1,ax=ax1, aspect=40,pad=0.08,shrink=0.8)
img2 = ax2.imshow(masked_NDVI_20070618, cmap='RdYlGn', vmin=-1, vmax=1)
ax2.set_title("NDVI_20070618")
cbar2 = fig.colorbar(img2,ax=ax2, aspect=40,pad=0.08,shrink=0.8)
img3 = ax3.imshow(masked_NDVI_20080805, cmap='RdYlGn', vmin=-1, vmax=1)
ax3.set_title("NDVI_20080805")
cbar3 = fig.colorbar(img3,ax=ax3, aspect=40,pad=0.08,shrink=0.8)
img4 = ax4.imshow(masked_NDVI_20081105, cmap='RdYlGn', vmin=-1, vmax=1)
ax4.set_title("NDVI_20081105")
cbar4 = fig.colorbar(img4,ax=ax4, aspect=40,pad=0.08,shrink=0.8)
img5 = ax5.imshow(masked_NDVI_20090508, cmap='RdYlGn', vmin=-1, vmax=1)
ax5.set_title("NDVI_20090508")
cbar5 = fig.colorbar(img5,ax=ax5, aspect=40,pad=0.08,shrink=0.8)
img6 = ax6.imshow(masked_NDVI_20100626, cmap='RdYlGn', vmin=-1, vmax=1)
ax6.set_title("NDVI_20100626")
cbar6 = fig.colorbar(img6,ax=ax6, aspect=40,pad=0.08,shrink=0.8)
img7 = ax7.imshow(masked_NDVI_20100926, cmap='RdYlGn', vmin=-1, vmax=1)
ax7.set_title("NDVI_20100926")
cbar7 = fig.colorbar(img7,ax=ax7, aspect=40,pad=0.08,shrink=0.8)
plt.show(fig)そうすると以下のような結果を得られます。
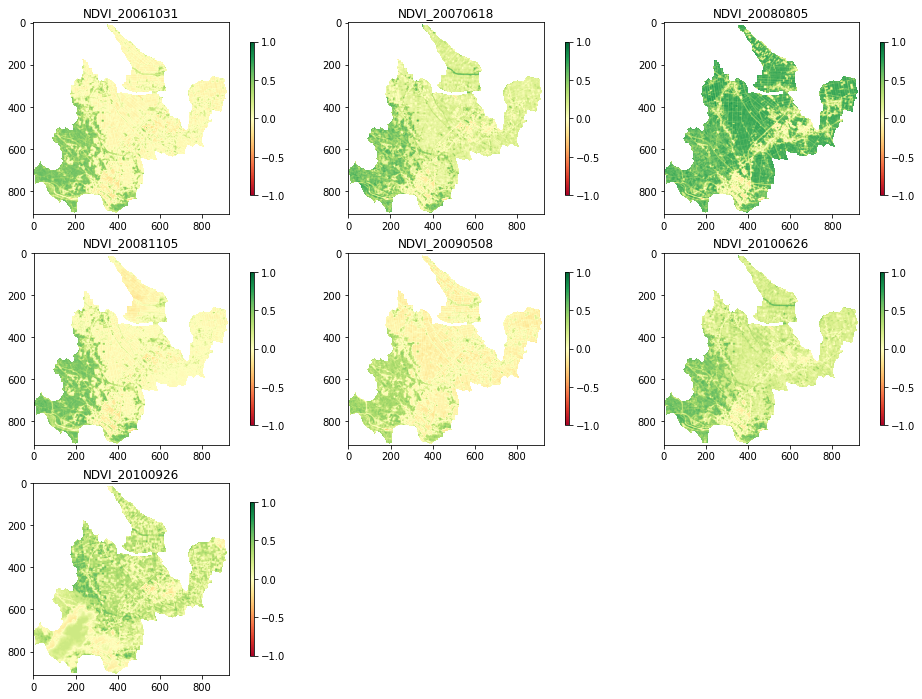
耕作が行われていない可能性のある圃場を抽出
ここまでで準備が整いました。いよいよ遊休農地、つまりは耕作が行われていない可能性のある圃場を抽出していきましょう。
これらの判定条件として、ここでは稲の作付けがされていない圃場、つまり周囲と比較してNDVI値の季節変動が少ない圃場や夏場のNDVI値が小さい圃場を判定のための簡易的な条件とします。
しかし、闇雲にシーン全体から上記の条件を当てはめてしまっても、圃場以外のエリアも大量に抽出されてしまうことが考えられます。そのため、まず初めにどこが圃場なのかを判定する必要があります。その上で、全ての時期のNDVIデータにおいてNDVIの変化が少ない圃場を抽出することとします。
圃場データとして、ここでは農林水産省が公開している筆ポリゴンデータを利用します※。これは圃場の一つ一つが単一のポリゴンとして格納されており、これを用いて、それぞれの圃場ごとのデータを抽出することが可能となります。
※なお、この筆ポリゴンが公開されているのは2020年以降のデータであるため、実際は衛星データの撮像日である2006年~2010年と一致しませんので、あくまで参考としての利用になります。
はじめに筆ポリゴンデータをGeoPandasのデータフレームとして読み込み、衛星画像と重畳するために投影変換を行います。
なお、GeoPandasって?という方は以下の記事をご覧ください。
地理空間情報を扱うなら知っておきたいPythonライブラリ、GeoPandas入門~基礎編~
## 筆ポリゴンの読み込み
## 藤崎町の筆ポリゴンを抽出
# 筆ポリゴンの保存先のフォルダを指定
# ※自身の環境に合わせて指定先は変更してください
POLYGON_PATH = "/content/drive/MyDrive/work/study/Sorabatake/06_fude_polygon/02青森県(2021公開)/02361藤崎町(2021公開)"
POLYGON_SHP = "02361藤崎町(2021公開)__aa.shp"
df_fude_polygon = gpd.read_file(os.path.join(POLYGON_PATH, POLYGON_SHP), encoding="shift-jis")
#データの確認
df_fude_polygon
Fujisaki_PaddyField = df_fude_polygon[df_fude_polygon["耕地の種類"].isin(["田"])]
Fujisaki_PaddyField = Fujisaki_PaddyField.drop(columns=["公開年度","調製年度"])
#投影変換
reprojest_Fujisaki_PaddyField = Fujisaki_PaddyField.to_crs({"init":"epsg:32654"})
reproject_PaddyFiled_out = os.path.join(POLYGON_PATH, "epsg_32654-"+ POLYGON_SHP)
#出力
reprojest_Fujisaki_PaddyField.to_file(driver="ESRI Shapefile",filename=reproject_PaddyFiled_out, encoding="shift-jis")
#抽出した藤崎町の筆ポリゴンを描画
f,ax = plt.subplots(1, figsize=(6,6))
ax = reprojest_Fujisaki_PaddyField.plot(ax=ax)
plt.show同じ場所のデータを取得している場合には、以下のような結果を得られます。広い範囲で田畑が広がっていることがわかりますね。
こちらで可視化した各ポリゴン内において、衛星画像から算出したNDVI値の平均を時系列に紐付けしていきます。
# 自身の環境に合わせて指定先は変更してください
MASK_DIR = "/content/drive/MyDrive/work/study/Sorabatake/03_ndvi/02_fujisaki_masked"
ndvi_list = os.listdir(MASK_DIR)
ndvi_list = [f for f in ndvi_list if os.path.isfile(os.path.join(MASK_DIR, f))]
print(ndvi_list)
## NDVI画像の読み込み
for ndvi_img in ndvi_list:
ndvi_path = os.path.join(MASK_DIR, ndvi_img)
ndvi_title = ndvi_img[-16:-8]
print(ndvi_path, ndvi_title)
#ポリゴン毎のndvi値の平均を格納
#画像毎にリスト初期化
ndvi_mean = []
print(str(ndvi_path) + " : 処理開始")
with rasterio.open(ndvi_path) as ndvi_src:
## 筆ポリゴンの読み込み
for index, item in reprojest_Fujisaki_PaddyField.iterrows():
masks = item.geometry
# 各筆ポリゴンに沿ってマスク処理
out_ndvi_img, out_transform = rasterio.mask.mask(ndvi_src, [masks], nodata=np.nan, crop=False)
# マスク内のNDVI値の平均を算出 -> リストへ
ndvi_mean.append(np.nanmean(out_ndvi_img))
#print(np.nanmean(out_ndvi_img))
#print("------------------")
# NDVI平均値のリストをdfに結合
reprojest_Fujisaki_PaddyField[ndvi_title] = ndvi_mean
print(str(ndvi_path) + "処理終了")
print("---結果の確認---")
reprojest_Fujisaki_PaddyField算出した結果に対して、グラフ呼び出しを簡単にするためにカラムの順番を入れ替えます。
## NDVI値の時系列をソート
recolums = ["id","耕地の種類","geometry", "20061031", "20070618",
"20080805", "20081105", "20090508", "20100626", "20100926"]
reprojest_Fujisaki_PaddyField = reprojest_Fujisaki_PaddyField.reindex(columns=recolums)
## dfを別名保存
reprojest_Fujisaki_PaddyField.to_file(driver='ESRI Shapefile',
filename = POLYGON_PATH + "/ndvi_clip.shp",
encoding="shift-jis")さて、算出した結果をグラフ化してみましょう。
## 追加したデータフレームからNDVI値を取得し、グラフ表示
# データの読み込み
# ※自身の環境に合わせて指定先は変更してください
ndvi_clip_path = "/content/drive/MyDrive/work/study/Sorabatake/06_fude_polygon/02青森県(2021公開)/02361藤崎町(2021公開)/ndvi_clip.shp"
ndvi_clip_df = gpd.read_file(ndvi_clip_path, encoding="shift-jis")
ndvi_clip_df_t = ndvi_clip_df.drop(columns=["耕地の種類", "geometry"])
ndvi_clip_df_t = ndvi_clip_df_t.T
list_tmp = ndvi_clip_df_t.loc["id"]
ndvi_clip_df_t = ndvi_clip_df_t.drop(index=["id"])
ndvi_clip_df_t.columns = list_tmp
ndvi_clip_df_t.plot.line(
figsize = (10, 6),
title = "NDVI time series change",
ylim = ([-1.0, 1.0]),
xlabel = "Date",
ylabel = "NDVI_value",
alpha=0.5,
legend = False)結果が表示されました。

ただし、このグラフだけではデータ量(対象ポリゴン数:6538個)が多すぎるため、細かいNDVIの変動が良くわからず、遊休農地を見つけ出すことができません。そこで、もう少しデータ数を絞って、傾向を見てみましょう。
# もう少しデータ数を減らしてみます
plot_item = []
# rangeの数字を変えることで表示データ数を変更可
for i in range(30):
plot_item.append(i)
ndvi_clip_df_t.plot.line(y = plot_item,
figsize = (10, 6),
title = "NDVI time series change",
ylim = ([-1.0, 1.0]),
xlabel = "Date",
ylabel = "NDVI_value",
alpha=0.5,
legend = False)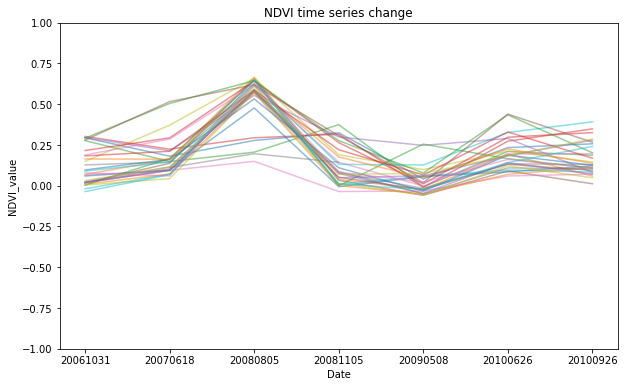
変化がわかりやすくなりました。
夏場になると植物が青々としていることからも想像できると思うのですが、農作物は夏場に向けてNDVIが高くなる傾向があります。そのような中で、夏場であってもNDVI値が高くならない区画があることも分かります。
そこで、NDVIが変化していない、つまりは耕作がされていない可能性のあるポリゴン(圃場)を絞ってみます。これらの圃場の特徴として、夏場でもNDVIの値が低いと仮定し、夏場である”20080805”のデータでNDVI値が0.0以下のポリゴンを抽出してみましょう。
## 耕作されていない可能性のあるデータを探す
## 耕作がされていない、つまり作付けが行われていないため夏場でもNDVIの値が低いと仮定
#ndvi_clip_df=ndvi_clip_df.drop(columns=["耕地の種類", "geometry"])
ndvi_low_id = ndvi_clip_df[ndvi_clip_df["20080805"]<= 0.0]
ndvi_low_idこのように夏場であってもNDVIの値が低い圃場がいくつか抽出されました。
実際に、NDVIが変化しない圃場を時系列に確認してみると、以下の通りでした。赤く囲っている範囲はたしかに目視でみても変化がなさそうな圃場に見えます。
実際にはこの場所が実際に耕作されていないのか、それ以外の理由でこのように見えているのかは定かではありませんが、少なくとも2006年から2010年までの間においてはNDVIが低い、つまり植生が比較的少ない状態であったことがデータから示唆されます。

このように衛星画像、特に赤外情報を含めた複数の波長をうまく組み合わせ、時系列に解析することで地上の様子をモニタリングし、場合によっては見たい対象物(今回の場合は作付けを行っていない田畑)を抽出することも可能となります。
コードはGITHUBでも公開しています。https://github.com/sorabatake/softwaredesign_1


