お米の食味ランク別に農地の様子を衛星データで比較してみた
お米の食味ランクで特Aに認定された地域と、認定されていない地域の圃場の様子の違いを衛星データを使って調査してみました。
はじめに
お米のおいしさの評価の一つとして、一般財団法人日本穀物検定協会が毎年3月に発表する「米の食味ランキング」があります。
食味ランキングでは、複数産地のコシヒカリのブレンド米を基準米とし、基準米よりも特に良好なものを「特A」、良好なものを「A」などとして発表されています。
下記は特Aランク一覧表で、どの地域のどの品種のお米が特Aに認定されたかがまとめられています。
https://www.kokken.or.jp/data/ranking_specialA.pdf
https://www.kokken.or.jp/data/ranking_rankbetu.pdf
以前宙畑では、農業における衛星データの利用方法の一つとして、おいしいお米の生産に衛星画像を活用している事例(https://sorabatake.jp/3699/)を紹介しました。
青森県で生産されているお米「青天の霹靂」では、衛星画像を利用して適切な収穫時期の予測や肥料の量の調整を行い、おいしいお米の生産を行っているとのことでした。
衛星データを利用しておいしいお米の生産を行っている例があるなら、おいしいお米の農場は衛星データから取得できる情報で違いがわかるかもしれません。
そこで、特Aとそうでない地域の農場の違いを衛星データから探ってみようと思います。
「青天の霹靂」に聞く!衛星データを用いた広大な稲作地帯の収穫時期予測
対象地域と使用するデータ
今回は新潟県を対象に調査を行います。
一般財団法人日本穀物検定協会の評価結果によると、お米の評価では新潟県は6つの地域に分けられており、下記のようにうち2地域が特A認定、残り4地域がA評価になっています。
いずれにしてもA評価以上なので、新潟県のお米は美味しいことが分かります。
特A:上越、魚沼
A:中越、下越、佐渡、岩船
今回はSentinel-2の光学画像を使って3つの正規化指標を求めて、その推移の違いを可視化してみます。
正規化指標とは、主に2つの波長の反射率の違いを利用して対象を観測するものです。
例えば、植物の活性度がわかるNDVI(Normalized Difference Vegetation Index)正規化植生指標、NDWI(Normalized Difference Water Index)正規化水指標などがあります。
上記のようなスペクトルの差異を用いた指標のことを総称してNDSI(Normalized Difference Spectral Index)と呼ぶこともあります。
その他の例や詳しい説明は、以前の記事(https://sorabatake.jp/5192/)で紹介されているのでぜひ読んでみてください。
課題に応じて変幻自在? 衛星データをブレンドして見えるモノ・コト #マンガでわかる衛星データ
今回は下記の3つの正規化指標を用います。
①NDVI(Normalized Difference Vegetation Index):緑植生の茂り具合を示す指標
②SAVI(Soil Adjust Vegitation index):背景土壌の影響を補正した植生指標
③緑と近赤外のNDSI(Normalized Difference Spectral Index):お米のタンパク質含有率が相関があるとされている指標

③は青天の霹靂の生産にも活用されている指標(https://www.rssj.or.jp/wp-content/uploads/2018/08/u08_64_h30.pdf)です。
お米のおいしさはタンパク質含有率と関係があるとされており、一般的にはタンパク質含有率が低い方が粘りの強いおいしいお米になります(低すぎるとむしろ評価が悪くなってしまう傾向もあるそう)。
新潟県の田んぼの筆ポリゴンを取得する
地域ごとの新潟県の田んぼのポリゴンデータを取得します。
まずは必要なライブラリをインポートします。
import pandas as pd
import warnings
import json
import os
warnings.filterwarnings('ignore')
# reminder that if you are installing libraries in a Google Colab instance you will be prompted to restart your kernal
try:
import geemap, ee
import seaborn as sns
import matplotlib.pyplot as plt
import geopandas as gpd
except ModuleNotFoundError:
if 'google.colab' in str(get_ipython()):
print("package not found, installing w/ pip in Google Colab...")
!pip install geemap seaborn matplotlib
!pip install geopandas
else:
print("package not found, installing w/ conda...")
!conda install mamba -c conda-forge -y
!mamba install geemap -c conda-forge -y
!conda install seaborn matplotlib -y
import geemap, ee
import seaborn as sns
import matplotlib.pyplot as plt
import geopandas as gpdGoogle Earth Engineの認証を行います。
コードを実行したら指示に従って認証を進めます。
try:
ee.Initialize()
except Exception as e:
ee.Authenticate()
ee.Initialize()まずは6つの地域のポリゴンデータを取得します。
ポリゴンデータとは、簡単に言えば緯度経度を持った形状のデータのことで、日本の市区町村ごとのポリゴンデータは国土交通省のサイト
(https://nlftp.mlit.go.jp/ksj/gml/datalist/KsjTmplt-N03-v3_0.html)
からダウンロードすることができます。サイトからダウンロードした新潟県の市区町村のポリゴンデータをGeoPandasで読み込みます。
fp = 'ダウンロードしたgeojsonのファイルパスを貼り付けてください。'
gdf = gpd.read_file(fp)
gdf
試しに取得したポリゴンデータを可視化してみます。
新潟県の形状を取得できていることがわかります。
gdf.plot()
上越、中越、下越、岩船、佐渡、魚沼の6地域の市区町村のリストを作成します。
地域区分は新潟県のホームページ(https://www.pref.niigata.lg.jp/site/link/link-sichoson.html)
を参考にしました。
Joetsu = ['上越市' ,'糸魚川市' ,'妙高市']
Tyuetsu = ['加茂市', '三条市', '長岡市','柏崎市', '小千谷市', '十日町市', '見附市', '田上町', '出雲崎町', '刈羽村']
Kaetsu = ['北区', '東区', '中央区', '江南区', '燕市', '秋葉区', '南区', '西区', '西蒲区', '新発田市', '燕市', '五泉市', '阿賀野市', '胎内市', '聖籠町', '弥彦村', '阿賀町']
Iwafune = ['村上市', '関川村', '粟島浦村']
Sado = ['佐渡市']
Uonuma = ['魚沼市', '南魚沼市', '湯沢町']地域ごとのGeoDataFrameを作成します。
取得した新潟県の市区町村のGeoDataFrameは”N03_004”列に市区町村名が格納されています。上で作成した各地域が含まれる行を抽出します。
Joetsu_gdf = gdf[gdf['N03_004'].isin(Joetsu)]
Tyuetsu_gdf = gdf[gdf['N03_004'].isin(Tyuetsu)]
Kaetsu_gdf = gdf[gdf['N03_004'].isin(Kaetsu)]
Iwafune_gdf = gdf[gdf['N03_004'].isin(Iwafune)]
Sado_gdf = gdf[gdf['N03_004'].isin(Sado)]
Uonuma_gdf = gdf[gdf['N03_004'].isin(Uonuma)]試しに魚沼のポリゴンデータを表示してみます。上のリストで指定した3つの市区町村の形状を抽出できていることがわかります。
Uonuma_gdf.plot()
筆ポリゴンを取得します。
筆ポリゴンとは圃場の一つ一つが単一のポリゴンとして格納されているもので、農林水産省(https://www.maff.go.jp/j/tokei/porigon/)のサイトで公開されています。
農林水産省では市区町村ごとに筆ポリゴンのjsonファイルを公開しています。 新潟県のjsonファイルを全てダウンロードして、一つのディレクトリにまとめて格納してください。
その後、下のコードでそれぞれのjsonファイルを一つのjsonにまとめます。
folder_path = 'jsonファイルをまとめたフォルダパスを貼り付けてください。'
df_fude_polygon = gpd.GeoDataFrame()
num = sum(os.path.isfile(os.path.join(folder_path, name)) for name in os.listdir(folder_path))
i = 0
firstLoop = True
for file in os.listdir(folder_path):
i += 1
print('{}/{}'.format(i, num))
fp = os.path.join(folder_path, file)
if firstLoop:
df_fude_polygon = gpd.read_file(fp)
firstLoop = False
else:
temp = gpd.read_file(fp)
df_fude_polygon = df_fude_polygon.append(temp)
outpath = os.path.join(folder_path, 'all.geojson')
df_fude_polygon.to_file(outpath, driver='GeoJSON', encoding='cp932')
df_fude_polygon# 一度all.geojsonを作成した後はこちらのコードでall.geojsonを読み込んでください。
# fp = os.path.join(out_path, 'all.geojson')
# df_fude_polygon = gpd.read_file(fp)筆ポリゴンには田んぼと畑のポリゴンデータが含まれています。
“land_type”のカラムの値が100のものが田んぼ、200のものが畑です。 今回は田んぼを対象に解析を行うため、”land_typeのカラムの値が100のものを抽出します。
Niigata_PaddyField = df_fude_polygon[df_fude_polygon["land_type"].isin([100])]
Niigata_PaddyField.head()
新潟県の田んぼのポリゴンデータを取得できました。
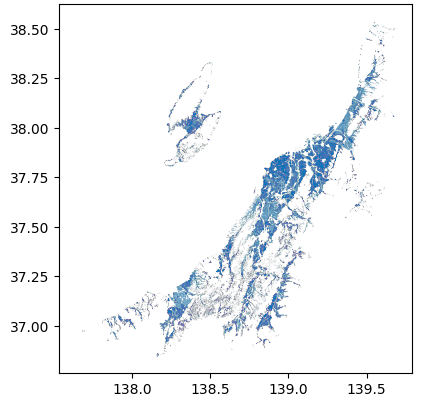
新潟県の田んぼで切り抜いた画像を表示する
取得した田んぼのポリゴンデータで切り抜いた画像を表示してみます。
新潟県の田んぼのポリゴンデータが格納されたGeoDataFrameをGEE用のee.Geometry.MultiPolygonに変換します。
新潟県全体では田んぼの数が非常に多く処理に時間がかかってしまうため、ここでは新潟市中央区の田んぼに絞って画像を表示してみます。
Joetsushi_gdf = gdf[gdf['N03_004']=='中央区']
features = []
aoi_gdf_PaddyField = gpd.sjoin(Niigata_PaddyField, Joetsushi_gdf, how="inner", op="within")
for i in range(len(aoi_gdf_PaddyField )):
x,y = aoi_gdf_PaddyField .iloc[i].geometry.exterior.coords.xy
coords = np.dstack((x,y)).tolist()
features.append(coords)
aoi_fude = ee.Geometry.MultiPolygon(features)Sentinel-2の2022年5月の画像の中から、被雲率が20%の画像の中央値画像を取得します。
GEEのSentinel-2の光学画像は、画素値の10000倍の値が格納されているため、”.divide(10000)”で10000で割ったものを取得します。※1
s2 = ee.ImageCollection('COPERNICUS/S2').filterDate('2022-05-01', '2022-5-31').filterBounds(aoi_fude).filter(ee.Filter.lt('CLOUDY_PIXEL_PERCENTAGE',70)).median().divide(10000)※1
各衛星、各バンドのスケールはGEEのサイトに記載されています。以下は今回の例です。
B2(青)、B3(緑)、B4(赤)を使ってTrueColor画像を作成します。
まずは田んぼのポリゴンデータで切り抜かずに画像を表示してみます。街や圃場の様子がよく観察できます。
color = ['FFFFFF', 'CE7E45', 'DF923D', 'F1B555', 'FCD163', '99B718',
'74A901', '66A000', '529400', '3E8601', '207401', '056201',
'004C00', '023B01', '012E01', '011D01', '011301']
pallete = {"min":0, "max":1, "bands": ['B4', 'B3', 'B2']}
map = geemap.Map(center=[37.885,139.08],zoom=14)
map.addLayer(s2, pallete, name='sentinel-2 TrueColor Image')
map.addLayerControl()
map
次に上で作成した新潟市中央区の田んぼのポリゴンデータ(aoi_fude)でclipします。
上の画像でスタジアム付近にあった圃場の形状を切り抜けていることがわかります。
color = ['FFFFFF', 'CE7E45', 'DF923D', 'F1B555', 'FCD163', '99B718',
'74A901', '66A000', '529400', '3E8601', '207401', '056201',
'004C00', '023B01', '012E01', '011D01', '011301']
pallete = {"min":0, "max":1, "bands": ['B4', 'B3', 'B2']}
map = geemap.Map(center=[37.885,139.08],zoom=14)
map.addLayer(s2.clip(aoi_fude), pallete, name='sentinel-2 TrueColor Image')
map.addLayerControl()
map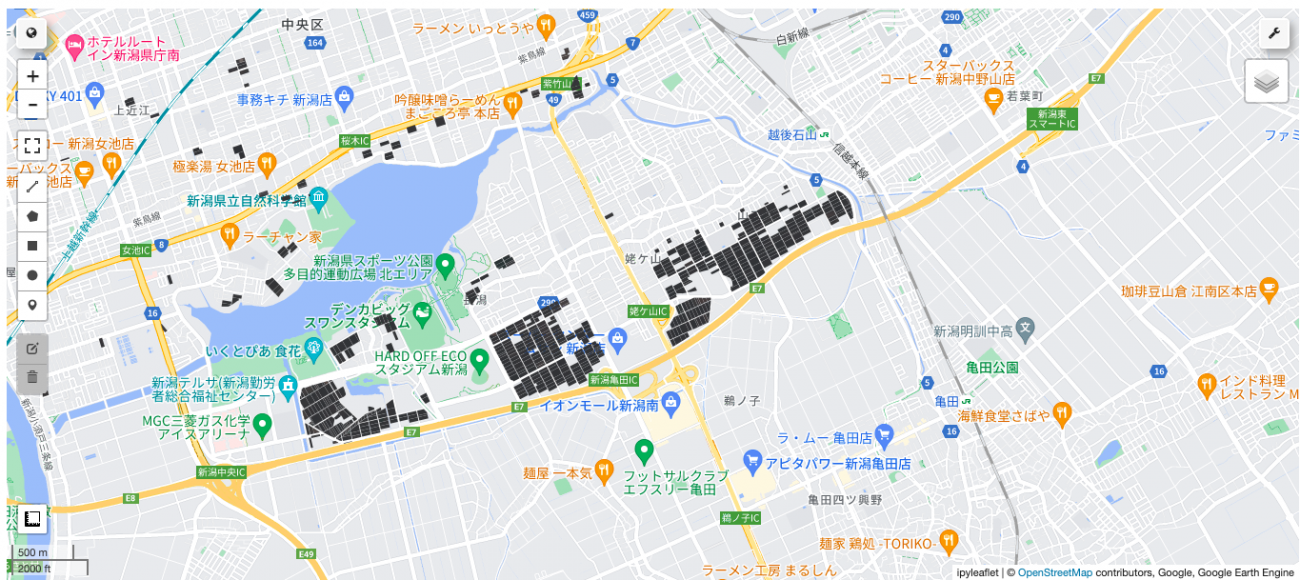
地域ごとの正規化指標の時系列変化を取得する
6つの地域ごとの正規化指標の時系列変化を取得して、推移の様子を比較します。
対象地域の田んぼのポリゴンデータを取得する関数を定義します。 全体の田んぼのポリゴンデータを含むGeoDataFrameと、対象地域のポリゴンデータを含むGeoDataFrameを引数として、対象地域の田んぼのポリゴンデータを返します。
田んぼのポリゴン数が多いとタイムアウトになってしまうため、今回は各地域5000区画をランダムに取得します。
※より効率の良いコードがあればタイムアウトにならないかもしれません。より良いコードがあれば教えていただけますと幸いです。
def get_aoi_fude(gdf_PaddyField, gdf_area):
features = []
aoi_gdf_PaddyField = gpd.sjoin(gdf_PaddyField, gdf_area, how="inner", op="within")
aoi_gdf_PaddyField = aoi_gdf_PaddyField.sample(n=5000, random_state=0, ignore_index=True)
for i in range(len(aoi_gdf_PaddyField)):
x,y = aoi_gdf_PaddyField.iloc[i].geometry.exterior.coords.xy
coords = np.dstack((x,y)).tolist()
features.append(coords)
aoi_fude = ee.Geometry.MultiPolygon(features)
return aoi_fude雲ピクセルのマスク処理を行う関数を定義します。今回使用するSentinel-2では”QA60”のバンドに雲の情報が格納されています。QA60はBit10とBit11にデータが存在しており、Bit10の値が1であれば雲あり、Bit11の値が1であれば巻雲ありとなっています。引数で与えられた画像から、QA60バンドで「Bit10が0かつBit11が0」となっているピクセル以外をマスクし、マスク画像を返します。
詳細はカタログページ(https://developers.google.com/earth-engine/datasets/catalog/COPERNICUS_S2_SR#description)を参照してください。
def cloudmask(img):
qa = img.select('QA60')
cloudmask = 1 << 10
cirrusmask = 1 << 11
mask = qa.bitwiseAnd(cloudmask).eq(0) and (qa.bitwiseAnd(cirrusmask).eq(0))
return img.updateMask(mask).divide(10000)今回扱う3つの正規化指標のNDVI、SAVI、緑と近赤外のNDSIを算出する関数を定義します。
# NDVI
def get_ndvi(img):
red = img.select('B4')
nir = img.select('B8')
numerator = nir.subtract(red)
denominator = nir.add(red)
ndvi = numerator.divide(denominator).rename("NDVI")
return ndvi
# SAVI = ((NIR - Red)/(NIR + Red + L)) * (1 + L)
def get_savi(img, L=0.5):
red = img.select('B4')
nir = img.select('B8')
numerator = nir.subtract(red)
denominator = nir.add(red).add(L)
savi = numerator.divide(denominator).multiply(L+1).rename("SAVI")
return savi
# NDSI_G-NIR = (NIR - Green) / (NIR + Green)
def get_ndsi_g_nir(img):
green = img.select('B3')
nir = img.select('B8')
numerator = nir.subtract(green)
denominator = nir.add(green)
mndwi = numerator.divide(denominator).rename("NDSI_G_NIR")
return mndwi各地域の正規化指標の時系列変化を取得します。 2022年の被雲率70%以下の画像を使用して、月ごとの正規化指標を取得します。 正規化指標は、上で取得した5000区画の田んぼでclipした画像の平均値を取得します。
def get_ndsi_list(aoi_gdf):
monthly_mean_ndvi_list_aoi = []
monthly_mean_savi_list_aoi = []
monthly_mean_ndsi_g_nir_list_aoi = []
geometry = get_aoi_fude(Niigata_PaddyField, aoi_gdf)
s2_without_cloud = ee.ImageCollection('COPERNICUS/S2').filterDate('2022-01-01', '2022-12-31').filterBounds(geometry).filter(ee.Filter.lt('CLOUDY_PIXEL_PERCENTAGE',70))
for month in months:
s2_mask = s2_without_cloud.filter(ee.Filter.calendarRange(month, month,'month')).map(cloudmask).median()
ndvi = get_ndvi(s2_mask)
savi = get_savi(s2_mask, L=0.5)
ndsi_g_nir = get_ndsi_g_nir(s2_mask)
monthly_mean_ndvi = ndvi.reduceRegion(reducer=ee.Reducer.mean(), geometry=geometry, scale=10)
monthly_mean_ndvi_list_aoi.append(monthly_mean_ndvi.get('NDVI').getInfo())
monthly_mean_savi = savi.reduceRegion(reducer=ee.Reducer.mean(), geometry=geometry, scale=10)
monthly_mean_savi_list_aoi.append(monthly_mean_savi.get('SAVI').getInfo())
monthly_mean_ndsi_g_nir = ndsi_g_nir.reduceRegion(reducer=ee.Reducer.mean(), geometry=geometry, scale=10)
monthly_mean_ndsi_g_nir_list_aoi.append(monthly_mean_ndsi_g_nir.get('NDSI_G_NIR').getInfo())
monthly_mean_ndvi_list.append(monthly_mean_ndvi_list_aoi)
monthly_mean_savi_list.append(monthly_mean_savi_list_aoi)
monthly_mean_ndsi_g_nir_list.append(monthly_mean_ndsi_g_nir_list_aoi)months = [1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12]
monthly_mean_ndvi_list = []
monthly_mean_savi_list = []
monthly_mean_ndsi_g_nir_list = []
aoi_gdf_list = [Joetsu_gdf, Tyuetsu_gdf, Kaetsu_gdf, Iwafune_gdf, Sado_gdf, Uonuma_gdf]
for aoi_gdf in aoi_gdf_list:
get_ndsi_list(aoi_gdf)正規化指標ごとにグラフを表示します。
plt.plot(months, monthly_mean_ndvi_list[0], label='Joetsu')
plt.plot(months, monthly_mean_ndvi_list[1], label='Tyetsu')
plt.plot(months, monthly_mean_ndvi_list[2], label='Kaetsu')
plt.plot(months, monthly_mean_ndvi_list[3], label='Iwafune')
plt.plot(months, monthly_mean_ndvi_list[4], label='Sado')
plt.plot(months, monthly_mean_ndvi_list[5], label='Uonuma')
plt.xlabel('Month')
plt.ylabel('Mean')
plt.title('Monthly Mean NDVI in 2022')
plt.legend()
plt.show()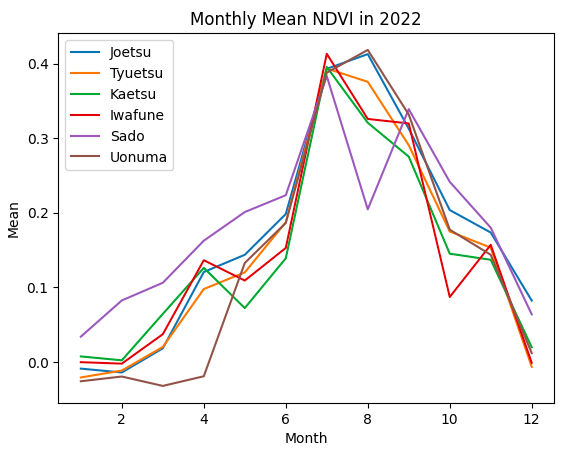
plt.plot(months, monthly_mean_savi_list[0], label='Joetsu')
plt.plot(months, monthly_mean_savi_list[1], label='Tyuetsu')
plt.plot(months, monthly_mean_savi_list[2], label='Kaetsu')
plt.plot(months, monthly_mean_savi_list[3], label='Iwafune')
plt.plot(months, monthly_mean_savi_list[4], label='Sado')
plt.plot(months, monthly_mean_savi_list[5], label='Uonuma')
plt.xlabel('Month')
plt.ylabel('Mean')
plt.title('Monthly Mean SAVI in 2022')
plt.legend()
plt.show()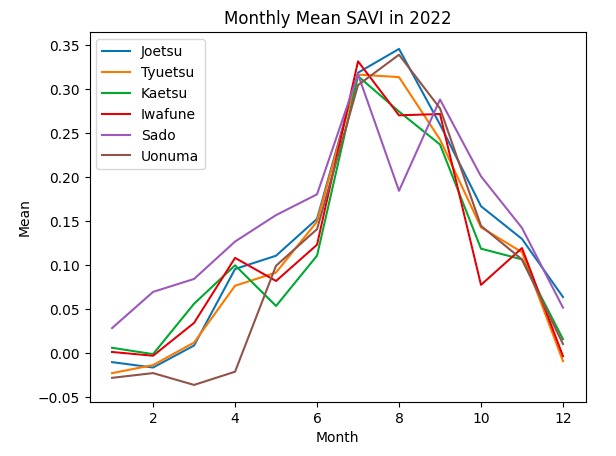
plt.plot(months, monthly_mean_ndsi_g_nir_list[0], label='Joetsu')
plt.plot(months, monthly_mean_ndsi_g_nir_list[1], label='Tyuetsu')
plt.plot(months, monthly_mean_ndsi_g_nir_list[2], label='Kaetsu')
plt.plot(months, monthly_mean_ndsi_g_nir_list[3], label='Iwafune')
plt.plot(months, monthly_mean_ndsi_g_nir_list[4], label='Sado')
plt.plot(months, monthly_mean_ndsi_g_nir_list[5], label='Uonuma')
plt.xlabel('Month')
plt.ylabel('Mean')
plt.title('Monthly Mean NDSI_G-NIR in 2022')
plt.legend()
plt.show()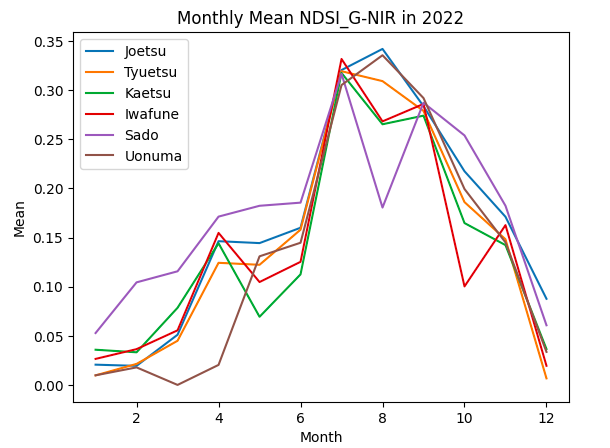
取得したグラフを見てみると、特Aである上越と魚沼では、どの正規化指標でも下記の特徴があると言えそうです。
・特Aの地域ではピークは8月、特Aでない地域は7月
・特Aの地域では、特Aでない地域に比べてピーク値が大きい
新潟県の食や文化に関して発信しているサイト(https://ichizen.online/niigataclip-blog/1293/)では、新潟県のお米の収穫時期は9月以降とのことです。
9月の正規化指標の値をみてみると、特Aと特Aでない地域は同程度の値となっています。 よって、特Aの地域の田んぼの特徴は「収穫直前にNDVI等がピーク値となり、そのピーク値が大きい。」と言えそうです。
また、上で述べたとおり、おいしいお米はタンパク質が少ないため、タンパク質と相関のある緑と近赤外のNDSIのピーク値は特Aの地域では小さい値を示すと考えていましたが、異なる結果となりました。収穫時期には同程度の値となっていることから、生育途中のピーク値は関係なく、収穫時期に低い値となれば良いと考えられます。
なお、今回はあくまで衛星データの分析結果を基に考察しているだけなので、新潟県は南北に長いために平均気温が異なるだろうこと、お米の適切な収穫時期は出穂後の日平均積算温度によって変わってくることなどから、特Aでない地域も適切なタイミングで収穫している可能性はあるかもしれません。
また、解析後に本記事を執筆する過程で「日照が不足し、充実が不良だと、相対的にタンパク質含量が高くなったり、粘りや硬さも変化する。そのため、日照が良い時期に出穂・登熟する良食味品種を選び、適切な栽培管理。豊作年の米が冷害年や日照不足の年の米に比べておいしい」といったタンパク質含有量以外の考慮すべきポイントが多くあることも分かりました。
まとめ
衛星データを使って、特Aのお米を生産している地域とそれ以外の地域での正規化指標の推移を調査してみました。
特Aの地域では、それ以外の地域と比較して推移の違いが見られ、「収穫直前時期にピーク値があり、その値は高い値となっている」ことが示唆されました。
今回は5000区画の田んぼで切り抜いた平均値を取得しました。区画をもっと増やしたり、全体の平均値ではなく、田んぼごとの推移を調べてみたりすると、より詳細な調査ができそうです。 また、無償公開されている衛星画像の中では解像度10mは高解像度ですが、商用衛星では5m以下のものもあります。より高解像度な衛星画像を使うことで新たな発見があるかもしれません。
衛星データを使った調査は、TellusやGoogle Earth Engineを使うことで初心者の方でも簡単に行うことができます。 今回の記事を参考にぜひ何らかの推移を調査してみてはいかがでしょうか。


